 Para Descargar PDF debe Abrir sesión.
Para Descargar PDF debe Abrir sesión.
Palabras clave: antibacterial agents, latin america, epidemiology
A partir de la observación de la actividad antimicrobiana a finales del siglo XIX y la producción a gran escala de los antibióticos en la década de 1940, los microorganismos han desarrollado múltiples mecanismos de resistencia a estos compuestos, debido a su uso indiscriminado y al paso de los años. Todo ello, dificulta el tratamiento de las enfermedades infecciosas. Este es el caso de varias especies de bacterias grampositivas y gramnegativas las cuales han disminuido su sensibilidad a los antibióticos ß-lactámicos como resultado del empleo inadecuado y abuso de su administración. Es por ello que la resistencia bacteriana a estos medicamentos representa un problema de salud pública que va en aumento en Latinoamérica, como consecuencia del surgimiento de cepas resistentes a varios de estos compuestos. Frente a estos antecedentes, y a que son escasos los análisis sistemáticos que se han efectuado sobre dicho tema, esta revisión bibliográfica tiene como objetivo resumir y analizar las publicaciones científicas sobre la resistencia a los antibióticos ß-lactámicos en algunos países de América Latina. Para esto se estudiaron publicaciones realizadas en los últimos cinco años e indexadas en las bases de datos PubMed, SciELO y LILACS-BIREME. Los resultados del análisis señalan que resulta común la identificación de genes de resistencia para los antibióticos ß-lactámicos en el medio ambiente, especialmente en el suelo y en las granjas de crianza de animales; y más del 40% de las cepas aisladas a partir de muestras clínicas presentan algún mecanismo de resistencia a esta familia de antibióticos.
|
Ideas claves
|
La primera observación de la actividad antimicrobiana ocurrió en el año de 1890, cuando Paul Vuillemin reportó antibiosis entre microorganismos[1]. Sin embargo, no fue sino hasta 1928, cuando Alexander Fleming evidenció la acción del primer antibiótico: la penicilina. Entre los años 1940 y 1950 se descubrió la mayor parte de sustancias antimicrobianas, las cuales son utilizadas hasta el presente[2]. Estos compuestos, además de ser utilizados para el tratamiento de las infecciones bacterianas, se emplean como inductores de crecimiento en las prácticas agrícolas y pecuarias[3],[4]. Estas actividades pueden afectar el desarrollo tanto de las células de especies animales como de vegetales[5],[6].
El hecho de que los antimicrobianos se encuentren en entornos ajenos al ambiente hospitalario contribuye a que surjan nuevas cepas resistentes[7]. Como consecuencia al empleo indiscriminado, y en ocasiones innecesario de antibióticos, la resistencia a los antimicrobianos se ha convertido en un problema de salud pública que va de la mano con el incremento en la morbimortalidad a nivel mundial[8]. Este hecho es relevante en países en vías de desarrollo que presentan los mayores índices de resistencia a estos compuestos en comparación a los países desarrollados[9]. Frente a este antecedente, nos planteamos como objetivo resumir y analizar las publicaciones científicas sobre la resistencia a los antibióticos ß-lactámicos en Latinoamérica. Para esto, en el presente trabajo describimos:
El levantamiento de la información se realizó usando las bases de datos PubMed, SciELO y LILACS-BIREME. De los 110 artículos publicados, entre enero de 2014 y febrero de 2019, utilizando como criterios de búsqueda “antibiotic resistance Latin America”, “antibiotic Latin America”, “beta lactam Latin America”, se analizaron 75 publicaciones científicas que estuvieron relacionadas con la resistencia a los antibióticos ß-lactámicos de Acinetobacter baumannii, Escherichia coli, Klebsiella pneumoniae, Pseudomonas aeruginosa, Staphylococcus aureus y Streptococcus pneumoniae. Los artículos que no trataron sobre la resistencia de estos microorganismos a esa familia de antibióticos no se consideraron para el análisis.
La actividad antimicrobiana de los agentes antibióticos se basa en la pérdida de estructuras celulares o la inhibición de procesos esenciales en la biología de la bacteria[10]. El amplio espectro de acción de estos agentes incluye la inhibición de la función y biosíntesis de la pared celular[10], la biosíntesis de proteínas[11], la traducción de proteínas[12], la transcripción del ADN[13] y el super enrollamiento del material genético[14].
Inhibición de la síntesis de la pared celular
En las bacterias, la pared celular se compone de biopolímeros, siendo esta indispensable para mantener la funcionalidad, integridad y forma celular[11]. El peptidoglucano, presente en la pared de las bacterias tanto grampositivas como gramnegativas, es una macromolécula constituida por redes de cadenas paralelas del disacárido N-acetilglucosamina: N-acetil murámico dispuestas a manera de malla a través de puentes peptídicos[15]. Esta estructura es indispensable para el ciclo de vida bacteriano, de ahí que es un blanco molecular ampliamente estudiado para la generación de antibióticos nuevos o modificados a partir de los ya existentes. Entre estos compuestos se encuentran los antibióticos ß-lactámicos como la penicilina, los carbapenémicos o las cefalosporinas, las cuales son capaces de inhibir a las proteínas de unión a la penicilina (Penicillin Binding Proteins, PBP), responsables de la formación de enlaces peptídicos, con lo cual se bloquea la formación de la red de peptidoglucano[16] (Figura 1).
Inhibición de la síntesis proteica
La traducción de ácido ribonucleico a proteína es un proceso celular primordial que depende de los ribosomas para el desarrollo de organismos eucariotas y procariotas. A pesar de la similitud de los procesos, los ribosomas procariotas poseen dos subunidades ribosomales 50S y 30S, los cuales difieren considerablemente de los ribosomas eucariotas que miden 60S y 40S. Esta diferencia básica entre las células procariotas y eucariotas ha sido explotada para generar antibióticos dirigidos específicamente a bacterias que permitan bloquear la traducción y síntesis de proteínas, lo cual resulta en el colapso del sistema funcional y estructural de la célula[17] (Figura 2).
Figura 2. Actividad de los antibióticos inhibidores de la síntesis de las proteínas.
Inhibición de la síntesis de material genético
El acceso a las diferentes estructuras celulares para llevar a cabo procesos moleculares-replicación, transcripción y reparación en el ADN depende de la topología (estructura) del mismo[18]. Las topoisomerasas son enzimas capaces de modificar la topología de las cadenas de ADN, por lo que su función es indispensable para la biología de la célula. Entre estas, la ADN girasa y topoisomerasa IV bacterianas resultan blancos para algunos antibióticos ya que bloquean el ciclo de vida del organismo[14]. Las fluoroquinolonas, por ejemplo inhiben a la ADN girasa de las bacterias gramnegativas y a la topoisomerasa IV de las grampositivas, mediante el bloqueo de la región de la enzima que se une al ADN generando cortes en la doble hélice del ADN y consecuentemente la lisis celular[19] (Figura 3).
Figura 3. Actividad de las fluoroquinolonas sobre la girasa.
Debido a la presión selectiva ejercida por los antibióticos, las bacterias desarrollan resistencia a estos, favoreciendo las bases genéticas que les confieren supervivencia por medio de:
Los mecanismos de resistencia son capaces de cambiar o modificar la biología de la bacteria para:
Para reducir o prevenir la entrada del antibiótico, las bacterias disminuyen la permeabilidad de la membrana extracelular generando menos canales de entrada (por ejemplo, porinas) o modificando los canales para hacerlos más selectivos. De la misma manera, la (sobre) producción de bombas de flujo le permite a la bacteria expulsar el antibiótico desde el espacio periplásmico hacia afuera de la célula por medio de transporte activo[22].
La modificación de la molécula diana del antibiótico es otro mecanismo que confiere resistencia, ya que las alteraciones genéticas o postranscripcionales de esta molécula pueden disminuir drásticamente su afinidad por el antibiótico. En consecuencia, puede bloquear la actividad del antimicrobiano[21].
Finalmente, la capacidad de las bacterias para modificar o inhibir a los antibióticos se ha visto directamente relacionada con la acción enzimática (por ejemplo enzimas hidrolíticas, acetiltransferasas, entre otras), las cuales catalizan cambios bioquímicos en el antimicrobiano[20]. El exhaustivo estudio de este método de resistencia ha permitido identificar enzimas capaces de degradar diferentes principios activos presentes en los antibióticos comúnmente usados como la penicilina, cefalosporinas y carbapenémicos[21].
Un ejemplo de estas enzimas son las ß-lactamasas que cada vez se identifican con mayor frecuencia en el nosocomio. Entre estas enzimas, las ß-lactamasas de espectro ampliado confieren a la bacteria resistencia a todos los ß-lactámicos, excepto al aztreonam o las carbapenemasas que les permite desarrollar resistentencia a los carbapenémicos. Esto representa un desafío para aplicar alternativas terapéuticas a las infecciones causadas por estos microorganismos[23].
Resistencia de los cocos grampositivos a los ß-lactámicos
Staphylococcus aureus es una especie de coco grampositivo, causante de infecciones locales y sistémicas[24],[25]. El aislamiento de cepas de Staphylococcus aureus resistentes a la meticilina representa un serio problema de salud pública a nivel mundial. Estas cepas se originan a causa de una mutación en la proteína Penicillin Binding Proteins 2a, lo cual les confiere resistencia a los antibióticos ß-lactámicos[26].
En Latinoamérica la principal cepa de Staphylococcus aureus resistentes a la meticilinacirculante es la USA300[27]. Si bien son comunes las infecciones hospitalarias por este microorganismo, la mortalidad no es significativamente superior respecto a las infecciones causadas por Staphylococcus aureus sensibles a meticilina[28].
En esta región la tasa de aislamientos de Staphylococcus aureus resistentes a la meticilina es considerable. Por ejemplo, este valor osciló entre 46% y 62% durante el año 2012, de un total de 981 cepas de Staphylococcus aureus identificadas en 17 centros médicos de seis países diferentes[29]. Lo que representa una señal de alerta respecto a la transmisión de este microorganismo.
Un factor que contribuye a la diseminación de las cepas Staphylococcus aureus resistentes a la meticilina, tanto dentro como fuera de América Latina es la migración. Es así como una variante de la cepa USA300, propia de Ecuador y Colombia, se ha asociado como la responsable de un brote en Norteamérica. A partir de pacientes portadores turistas que habrían visitado esos dos países andinos[30].
Frente a esta situación, se han realizado varios esfuerzos con el propósito de encontrar nuevos fármacos para el tratamiento de las infecciones causadas por Staphylococcus aureus resistentes a la meticilina. Una alternativa terapéutica es la ceftarolina, una cefalosporina capaz de unirse a la Penicillin Binding Proteins 2a mutada e impedir la formación de la pared celular bacteriana[31].
Como parte del estudio realizado en 2012, Evaluación de la Resistencia a los Antimicrobianos (Assesing Antimicrobial Resistance Evaluation, AWARE), el 83,3% de las cepas Staphylococcus aureus resistentes a la meticilina fueron sensibles a la ceftarolina con un valor de concentración inhibitoria mínima al 90% de las cepas evaluadas de dos microgramos por mililitro[32].
Otra alternativa terapéutica es la tigecilina, una glicilclina que inhibe la síntesis de las proteínas bacterianas[33]. Más del 99% de las cepas Staphylococcus aureus resistentes a la meticilina aisladas en Latinoamérica fueron sensibles a este antibiótico con una concentración inhibitoria mínima al 90% de entre 0,06 microgramos por mililitro y 0,25 microgramos por mililitro[34],[35].
Además de este antimicrobiano, el glucopéptido telavancina, que actúa inhibiendo la transpeptidización durante la síntesis del peptidoglucano[36], ha mostrado tener una concentración inhibitoria mínima al 50% de 0,06 microgramos por mililitro en cepas Staphylococcus aureus resistentes a la meticilina[37].
Por otro lado, Streptococcus pneumoniae también es una especie de gran importancia epidemiológica. Esta bacteria es responsable de cuadros de meningitis, otitis media y neumonía, tanto en niños como en adultos[38]. Este coco grampositivo en la actualidad ha desarrollado resistencias a algunos antibióticos, los cuales son considerados como alternativas terapéuticas[39].
En Latinoamérica el serotipo más común de Streptococcus pneumoniae es el 19A. Datos epidemiológicos han demostrado que entre el año 2011 y 2014, el 12,8% de las cepas fueron resistentes a penicilina34. Por ejemplo, en México y Venezuela la resistencia a este antimicrobiano se demostró con una concentración inhibitoria mínima mayor o igual a cuatro microgramos por mililitro[40].
En Perú, de un total de 212 cepas asiladas a partir de niños menores de cinco años, que presentaban enfermedad neumocócica invasiva, el 82% de las cepas (n = 173) manifestaron al menos un mecanismo genético de resistencia. De las cuales, el 34% (n = 72) fueron resistentes a múltiples fármacos (del inglés, Multi Drug Resistant, MDR)[41].
Al igual que las cepas de Staphylococcus aureus resistentes a la meticilina una alternativa terapéutica para Streptococcus pneumoniae resistente a los ß-lactámicos en la región, es la tigeciclina que ha mostrado tener una concentración inhibitoria mínima al 90% de las cepas evaluadas de 0,06 microgramos por mililitro[35].
Resistencia de los bacilos gramnegativos a los ß-lactámicos
Entre los bacilos gramnegativos con mayores índices de resistencia a los antimicrobianos se encuentran las enterobacterias, Pseudomonas aeruginosa y Acinetobacter baumannii. Estos microorganismos son de alto interés epidemiológico, debido a la severidad de las infecciones que pueden causar y a las pocas alternativas terapéuticas[42].
Desde 2005 la prevalencia de cepas productoras de ß-lactamasas de espectro ampliado de origen nosocomial ha incrementado progresivamente en América Latina. Es así, que se han aislado cepas de Escherichia coli y de Klebsiella pneumoniae resistentes a las cefalosporinas de tercera generación en una proporción de entre 11 a 25% y del 45 al 53%, respectivamente[43].
Como parte de los análisis realizados en el Programa de Evaluación y Pruebas de Seguimiento de la Tigeciclina (Tigecycline Evaluation and Surveillance Trial, TEST), el 24,5% de las cepas de Escherichia coli aisladas en Latinoamérica fueron productoras de ß-lactamasas de espectro ampliado[44]. Gran parte de estas han sido aisladas a partir de urocultivos, como es el caso de Colombia[45]. No obstante, estos datos varían entre países. Por ejemplo, en Paraguay, en un estudio efectuado en 2012 durante tres meses, la incidencia de cepas productoras de ß-lactamasas de espectro ampliado de Escherichia coli y de Klebsiella pneumoniae fue de 6,6% y de 30,8%, respectivamente[46]
La producción de dichas enzimas por parte de estas bacterias ha contribuido a que el 50% de las cepas de Escherichia coli que se aíslan en la región sean resistentes a múltiples fármacos. A diferencia de otras localizaciones, como en Norteamérica, donde el porcentaje de cepas con esta característica no supera el 6%[44].
A parte de Escherichia coli, la enterobacteria Klebsiella pneumoniae también ha mostrado altos índices de resistencia. Los resultados del Estudio para el Seguimiento de las Tendencias de Resistencia antimicrobiana (Study for Monitoring Antimicrobial Resistance Trends, SMART), realizado con un total de 516 cepas de dicho microorganismo, mostró que 71,7% (n= 370) de ellas fueron portadoras de genes de ß-lactamasas de espectro ampliado, siendo los más comunes: blaCTX-M-5, blaCTX-M-2 y blaSHV-12[47]. En relación con los genes se ha visto que, a parte de los mencionados, también están presentes en la región blaCTX-M-2, blaCTX-M-59, blaTEM-15, blaSHV-45 y blaSHV-55[48].
Durante el bienio 2013-2015, la prevalencia de cepas de Klebsiella pneumoniae productoras de ß-lactamasas de espectro ampliado aisladas de infecciones intraabdominales y de pacientes de cuidados intensivos, fue de 42 y 46%, respectivamente[49].
En cuanto a los microorganismos productores de carbapenemasas, además de las enterobacterias[44], resulta de especial interés la bacteria Acinetobacter baumannii. Este microorganismo medioambiental se identifica frecuentemente en el suelo[50] y en el ambiente hospitalario[51]. Debido a la resistencia a los antibióticos carbapenémicos, la Organización Mundial de la Salud (OMS) lo ha clasificado como un agente infeccioso de gran importancia epidemiológica[52].
Latinoamérica es una de las regiones con mayores índices de resistencia de Acinetobacter baumannii a este grupo de antimicrobianos. Argentina, Colombia y Brasil son los países que mayor incidencia de cepas resistentes han reportado. En aproximadamente 20 años, el incremento de resistencia de este microorganismo en la región al imipenem ha aumentado hasta en 85%[49],[53].
A partir del primer reporte de una cepa de Pseudomonas aeruginosa portadora del gen blaKPC-3 en Colombia[54], la presencia de genes de resistencia a los carbapenémicos ha incrementado. Así, en Latinoamérica se han identificado varios genes como por ejemplo, blaKPC-2 tanto en enterobacterias como en Pseudomonas aeruginosa[55].
Para el caso de Acinetobacter baumannii, recientemente se ha notificado la presencia de los genes, blaOXA-23, blaOXA-51, y blaOXA-58, blaOXA-72 en cepas aisladas de pacientes hospitalizados[52],[56]. La alta diseminación de estos genes de resistencia se ve favorecida por plásmidos[57], lo que facilitaría la propagación nosocomial de las cepas resistentes.
El problema de la diseminación horizontal de los genes de resistencia ha contribuido a que varias cepas adquieran resistencia a diversos grupos de antimicrobianos, tal como sucede tanto a los carbapenémicos y a los ß-lactámicos. Esto se ha evidenciado en enterobacterias que pueden contener determinantes genéticos de resistencia a estos dos grupos de antimicrobianos[47], haciendo que el tratamiento de las infecciones causadas por estas cepas resistentes a múltiples fármacos se torne más complicado[44].
Además de la transferencia horizontal de genes de resistencia en los nosocomios, las actividades agrícolas y la crianza de animales pueden contribuir al surgimiento de cepas resistentes[4],[50],[58],[59],[60]. Por ejemplo, en las granjas avícolas de Brasil, Colombia, Ecuador y Perú se han identificado genes responsables por la expresión de las enzimas ß-lactamasas de espectro ampliado[61],[62],[63],[64],[65],[66],[67],[68],[69], lo que representa un problema sanitario.
Ante la alta prevalencia de cepas resistentes, se han buscado nuevas alternativas terapéuticas para las infecciones. La colistina, un desestabilizador de la membrana citoplasmática que se encontraba sin aplicación, es una de ellas. Sin embargo, en 2016 se reportó por primera vez el aislamiento de cepas de Escherichia coli portadoras del gen mcr-1, con concentración inhibitoria mínimas de entre cuatro microgramos por mililitro y 16 microgramos por mililitro en Argentina[70].
A partir de este primer reporte, además de Argentina, el gen mcr-1 también se ha identificado en cepas de enterobacterias en Brasil, Colombia, Ecuador y Venezuela. Asimismo, en varios casos estas cepas son productoras de ß-lactamasas de espectro ampliado y de carbapenemasas. La diseminación de estos genes de resistencia puede verse favorecida por la facilidad de desplazamiento. Por ejemplo, en 2016 se reportó la identificación de dos cepas de Escherichia coli productoras de ß-lactamasas de espectro ampliado y portadoras del gen mcr-1 a partir de dos viajeros no relacionados que visitaron Perú y Bolivia, lo cual pone en alerta a los sistemas de salud de la región[71].
Latinoamérica es una de las regiones del planeta en donde la resistencia a los antimicrobianos se ha incrementado considerablemente. Por ello, tal como señala nuestro análisis, la diseminación de genes de resistencia entre los microorganismos y de las cepas resistentes a múltiples fármacos representa un tema de interés público. El ambiente hospitalario, los viajes de portadores asintomáticos y las prácticas agropecuarias constituyen potenciales mecanismos de diseminación de microorganismos resistentes a múltiples fármacos portadores de genes de resistencia de ß-lactamasas de espectro ampliado y/o de carbapenemasas y consecuentemente provocar infecciones nosocomiales y comunitarias de difícil tratamiento.
Contribuciones
LBF, ZLAC y GME contribuyeron equitativamente en: la conceptualización, el análisis formal, en la escritura y en la edición dl manuscrito.
Agradecimientos
Los autores expresan su agradecimiento a la Dirección de Investigación y Desarrollo de la Universidad Técnica de Ambato por el financiamiento de sus salarios.
Conflictos de intereses
Los autores declaran no tener ningún conflicto de interés con los datos publicados.
Financiamiento
Este trabajo ha sido autofinanciado por los autores.
Material suplementario
Base de datos disponible en: https://figshare.com/articles/Database_xlsx/9922247

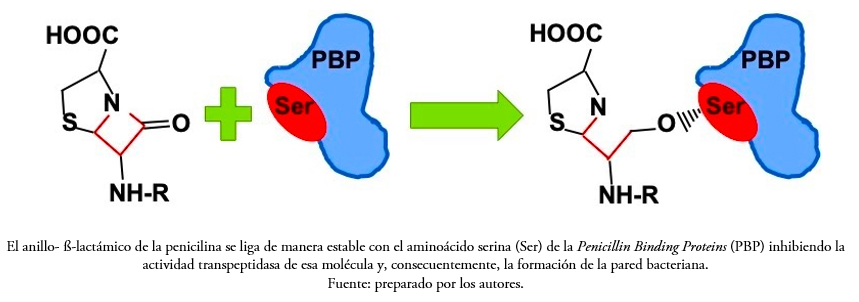 Figura 1. Actividad de los antibióticos ß-lactámicos sobre las proteínas de unión a las penicilinas (PBP´s).
Figura 1. Actividad de los antibióticos ß-lactámicos sobre las proteínas de unión a las penicilinas (PBP´s).

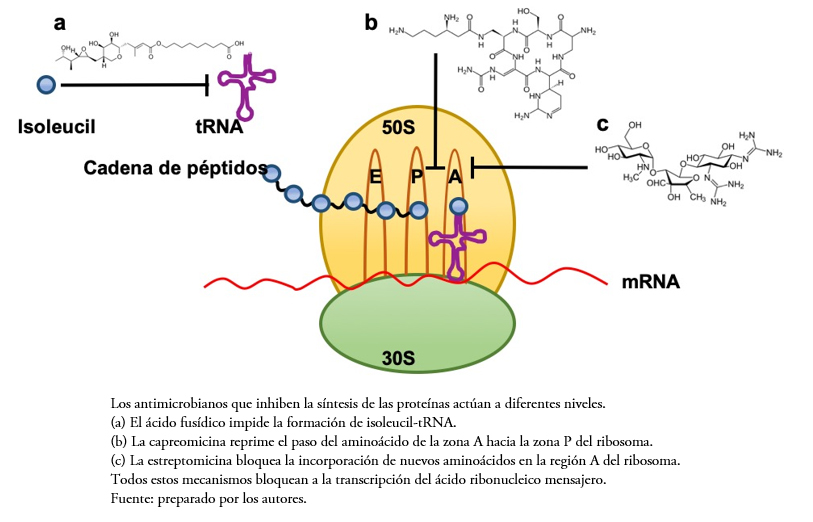 Figura 2. Actividad de los antibióticos inhibidores de la síntesis de las proteínas.
Figura 2. Actividad de los antibióticos inhibidores de la síntesis de las proteínas.

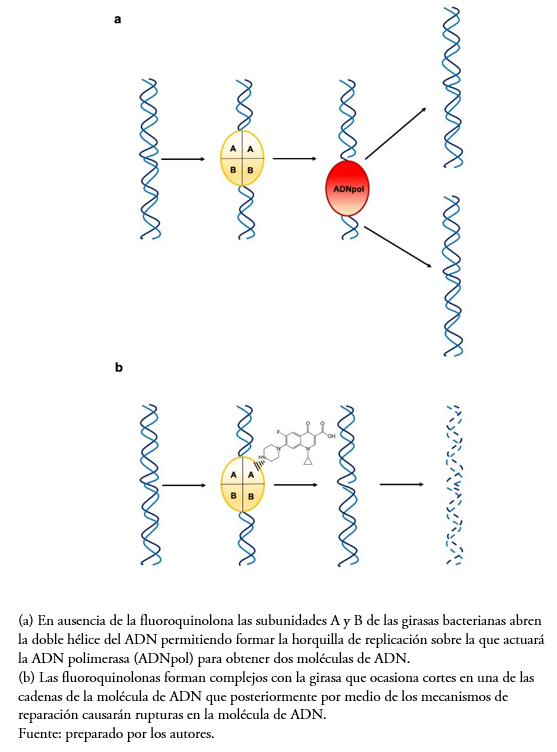 Figura 3. Actividad de las fluoroquinolonas sobre la girasa.
Figura 3. Actividad de las fluoroquinolonas sobre la girasa.
 Esta obra de Medwave está bajo una licencia Creative Commons Atribución-NoComercial 3.0 Unported. Esta licencia permite el uso, distribución y reproducción del artículo en cualquier medio, siempre y cuando se otorgue el crédito correspondiente al autor del artículo y al medio en que se publica, en este caso, Medwave.
Esta obra de Medwave está bajo una licencia Creative Commons Atribución-NoComercial 3.0 Unported. Esta licencia permite el uso, distribución y reproducción del artículo en cualquier medio, siempre y cuando se otorgue el crédito correspondiente al autor del artículo y al medio en que se publica, en este caso, Medwave.

Ever since antimicrobial activity was observed at the end of the XIX century and antibiotics were produced on a large scale in the ’40s, microorganisms have developed multiple resistance mechanisms, making treatment of infectious diseases difficult. For instance, several Gram-positive and Gram-negative bacteria lowered their sensitivity to ß-Lactam antibiotics as a result of their inadequate use and abuse. For this reason, microbial resistance to these drugs represents an increasing health problem in Latin America due to the emergence of drug-resistant bacterial strains. This review aims to summarize and analyze scientific literature reporting resistance to ß-lactam antibiotics in Latin America. We compiled scientific papers published during the last five years from PubMed, SciELO, and LILACS-BIREME. We found that: (i) it is common to identify resistance genes for ß-lactams in the soil and animal farms, and (ii) over 40% of strains isolated from clinical samples developed resistance against ß-lactam antibiotics.
 Autores:
María Fernanda Latorre-Barragan[1,2], Andrea Cristina Zurita-Leal[3], Marco Esteban Gudiño Gomezjurado[4]
Autores:
María Fernanda Latorre-Barragan[1,2], Andrea Cristina Zurita-Leal[3], Marco Esteban Gudiño Gomezjurado[4]

Citación: Latorre-Barragan MF, Zurita-Leal AC, Gudiño Gomezjurado ME. ß-lactam antibiotics resistance in Latin American countries. Medwave 2019;19(10):e7729 doi: 10.5867/medwave.2019.10.7729
Fecha de envío: 29/7/2019
Fecha de aceptación: 21/10/2019
Fecha de publicación: 20/11/2019
Origen: No solicitado
Tipo de revisión: Revisado por cuatro pares revisores externos a doble ciego

Nos complace que usted tenga interés en comentar uno de nuestros artículos. Su comentario será publicado inmediatamente. No obstante, Medwave se reserva el derecho a eliminarlo posteriormente si la dirección editorial considera que su comentario es: ofensivo en algún sentido, irrelevante, trivial, contiene errores de lenguaje, contiene arengas políticas, obedece a fines comerciales, contiene datos de alguna persona en particular, o sugiere cambios en el manejo de pacientes que no hayan sido publicados previamente en alguna revista con revisión por pares.
Aún no hay comentarios en este artículo.
Para comentar debe iniciar sesión
 Medwave publica las vistas HTML y descargas PDF por artículo, junto con otras métricas de redes sociales.
Medwave publica las vistas HTML y descargas PDF por artículo, junto con otras métricas de redes sociales.
 Nicolaou KC, Rigol S. A brief history of antibiotics and select advances in their synthesis. J Antibiot (Tokyo). 2018 Feb;71(2):153-184. | CrossRef | PubMed |
Nicolaou KC, Rigol S. A brief history of antibiotics and select advances in their synthesis. J Antibiot (Tokyo). 2018 Feb;71(2):153-184. | CrossRef | PubMed | Zaman SB, Hussain MA, Nye R, Mehta V, Mamun KT, Hossain N. A Review on Antibiotic Resistance: Alarm Bells are Ringing. Cureus. 2017 Jun 28;(6):e1403.
| CrossRef | PubMed |
Zaman SB, Hussain MA, Nye R, Mehta V, Mamun KT, Hossain N. A Review on Antibiotic Resistance: Alarm Bells are Ringing. Cureus. 2017 Jun 28;(6):e1403.
| CrossRef | PubMed | Jaramillo-Jaramillo AS, Cobo-Ángel CG, Moreno-Tolosa Y, Ceballos-Márquez A, Jaramillo-Jaramillo AS, Cobo-Ángel CG, et al. Resistencia antimicrobiana de Streptococcus agalactiae de origen humano y bovino. CES Med Vet y Zootec. 2018;13(1):62-79. | Link |
Jaramillo-Jaramillo AS, Cobo-Ángel CG, Moreno-Tolosa Y, Ceballos-Márquez A, Jaramillo-Jaramillo AS, Cobo-Ángel CG, et al. Resistencia antimicrobiana de Streptococcus agalactiae de origen humano y bovino. CES Med Vet y Zootec. 2018;13(1):62-79. | Link | Zareifopoulos N, Panayiotakopoulos G. Neuropsychiatric Effects of Antimicrobial Agents. Clin Drug Investig. 2017 May;37(5):423-437. | CrossRef | PubMed |
Zareifopoulos N, Panayiotakopoulos G. Neuropsychiatric Effects of Antimicrobial Agents. Clin Drug Investig. 2017 May;37(5):423-437. | CrossRef | PubMed | Gudiño ME, Blanco-Touriñán N, Arbona V, Gómez-Cadenas A, Blázquez MA, Navarro-García F. β-Lactam Antibiotics Modify Root Architecture and Indole Glucosinolate Metabolism in Arabidopsis thaliana. Plant Cell Physiol. 2018 Oct 1;59(10):2086-2098. | CrossRef | PubMed |
Gudiño ME, Blanco-Touriñán N, Arbona V, Gómez-Cadenas A, Blázquez MA, Navarro-García F. β-Lactam Antibiotics Modify Root Architecture and Indole Glucosinolate Metabolism in Arabidopsis thaliana. Plant Cell Physiol. 2018 Oct 1;59(10):2086-2098. | CrossRef | PubMed | Martínez JL. Antibiotics and Antibiotic Resistance Genes in Natural Environments. Science (80- ). 2008;321(5887):365–7. | CrossRef |
Martínez JL. Antibiotics and Antibiotic Resistance Genes in Natural Environments. Science (80- ). 2008;321(5887):365–7. | CrossRef | Sabtu N, Enoch DA, Brown NM. Antibiotic resistance: what, why, where, when and how? Br Med Bull. 2015;116:105-13. | CrossRef | PubMed |
Sabtu N, Enoch DA, Brown NM. Antibiotic resistance: what, why, where, when and how? Br Med Bull. 2015;116:105-13. | CrossRef | PubMed | Chokshi A, Sifri Z, Cennimo D, Horng H. Global Contributors to Antibiotic Resistance. J Glob Infect Dis. 2019 Jan-Mar;11(1):36-42. | CrossRef | PubMed |
Chokshi A, Sifri Z, Cennimo D, Horng H. Global Contributors to Antibiotic Resistance. J Glob Infect Dis. 2019 Jan-Mar;11(1):36-42. | CrossRef | PubMed | Epand RM, Walker C, Epand RF, Magarvey NA. Molecular mechanisms of membrane targeting antibiotics. Biochim Biophys Acta. 2016 May;1858(5):980-7. | CrossRef | PubMed |
Epand RM, Walker C, Epand RF, Magarvey NA. Molecular mechanisms of membrane targeting antibiotics. Biochim Biophys Acta. 2016 May;1858(5):980-7. | CrossRef | PubMed | Zhou P, Zhao J. Structure, inhibition, and regulation of essential lipid A enzymes. Biochim Biophys Acta Mol Cell Biol Lipids. 2017 Nov;1862(11):1424-1438. | CrossRef | PubMed |
Zhou P, Zhao J. Structure, inhibition, and regulation of essential lipid A enzymes. Biochim Biophys Acta Mol Cell Biol Lipids. 2017 Nov;1862(11):1424-1438. | CrossRef | PubMed | McCoy LS, Xie Y, Tor Y. Antibiotics that target protein synthesis. Wiley Interdiscip Rev RNA. 2011 Mar-Apr;2(2):209-32. | CrossRef | PubMed |
McCoy LS, Xie Y, Tor Y. Antibiotics that target protein synthesis. Wiley Interdiscip Rev RNA. 2011 Mar-Apr;2(2):209-32. | CrossRef | PubMed | Temiakov D, Zenkin N, Vassylyeva MN, Perederina A, Tahirov TH, Kashkina E, et al. Structural basis of transcription inhibition by antibiotic streptolydigin. Mol Cell. 2005 Sep 2;19(5):655-66. | PubMed |
Temiakov D, Zenkin N, Vassylyeva MN, Perederina A, Tahirov TH, Kashkina E, et al. Structural basis of transcription inhibition by antibiotic streptolydigin. Mol Cell. 2005 Sep 2;19(5):655-66. | PubMed | Ehmann DE, Lahiri SD. Novel compounds targeting bacterial DNA topoisomerase/DNA gyrase. Curr Opin Pharmacol. 2014 Oct;18:76-83. | CrossRef | PubMed |
Ehmann DE, Lahiri SD. Novel compounds targeting bacterial DNA topoisomerase/DNA gyrase. Curr Opin Pharmacol. 2014 Oct;18:76-83. | CrossRef | PubMed | Romaniuk JAH, Cegelski L. Bacterial cell wall composition and the influence of antibiotics by cell-wall and whole-cell NMR. Philos Trans R Soc B Biol Sci. 2015;370(1679). | CrossRef |
Romaniuk JAH, Cegelski L. Bacterial cell wall composition and the influence of antibiotics by cell-wall and whole-cell NMR. Philos Trans R Soc B Biol Sci. 2015;370(1679). | CrossRef | Cho H, Uehara T, Bernhardt TG. Beta-lactam antibiotics induce a lethal malfunctioning of the bacterial cell wall synthesis machinery. Cell. 2014 Dec 4;159(6):1300-11. | CrossRef | PubMed |
Cho H, Uehara T, Bernhardt TG. Beta-lactam antibiotics induce a lethal malfunctioning of the bacterial cell wall synthesis machinery. Cell. 2014 Dec 4;159(6):1300-11. | CrossRef | PubMed | Etebu E, Arikekpar I. Antibiotics: Classification and mechanisms of action with emphasis on molecular perspectives. Int J Appl Microbiol Biotechnol Res. 2016;4:90-101.
| Link |
Etebu E, Arikekpar I. Antibiotics: Classification and mechanisms of action with emphasis on molecular perspectives. Int J Appl Microbiol Biotechnol Res. 2016;4:90-101.
| Link | Nagaraja V, Godbole AA, Henderson SR, Maxwell A. DNA topoisomerase I and DNA gyrase as targets for TB therapy. Drug Discov Today. 2017 Mar;22(3):510-518. | CrossRef | PubMed |
Nagaraja V, Godbole AA, Henderson SR, Maxwell A. DNA topoisomerase I and DNA gyrase as targets for TB therapy. Drug Discov Today. 2017 Mar;22(3):510-518. | CrossRef | PubMed | van Eijk E, Wittekoek B, Kuijper EJ, Smits WK. DNA replication proteins as potential targets for antimicrobials in drug-resistant bacterial pathogens. J Antimicrob Chemother. 2017 May 1;72(5):1275-1284. | CrossRef | PubMed |
van Eijk E, Wittekoek B, Kuijper EJ, Smits WK. DNA replication proteins as potential targets for antimicrobials in drug-resistant bacterial pathogens. J Antimicrob Chemother. 2017 May 1;72(5):1275-1284. | CrossRef | PubMed | Giedraitienė A, Vitkauskienė A, Naginienė R, Pavilonis A. Antibiotic resistance mechanisms of clinically important bacteria. Medicina (Kaunas). /2011;47(3):137-46. | CrossRef | PubMed |
Giedraitienė A, Vitkauskienė A, Naginienė R, Pavilonis A. Antibiotic resistance mechanisms of clinically important bacteria. Medicina (Kaunas). /2011;47(3):137-46. | CrossRef | PubMed | Blair JM, Webber MA, Baylay AJ, Ogbolu DO, Piddock LJ. Molecular mechanisms of antibiotic resistance. Nat Rev Microbiol. 2015 Jan;13(1):42-51. | CrossRef | PubMed |
Blair JM, Webber MA, Baylay AJ, Ogbolu DO, Piddock LJ. Molecular mechanisms of antibiotic resistance. Nat Rev Microbiol. 2015 Jan;13(1):42-51. | CrossRef | PubMed | Masi M, Réfregiers M, Pos KM, Pagès JM. Mechanisms of envelope permeability and antibiotic influx and efflux in Gram-negative bacteria. Nat Microbiol. 2017 Feb 22;2:17001. | CrossRef | PubMed |
Masi M, Réfregiers M, Pos KM, Pagès JM. Mechanisms of envelope permeability and antibiotic influx and efflux in Gram-negative bacteria. Nat Microbiol. 2017 Feb 22;2:17001. | CrossRef | PubMed | Lakhundi S, Zhang K. Methicillin-Resistant Staphylococcus aureus: Molecular Characterization, Evolution, and Epidemiology. Clin Microbiol Rev. 2018 Sep 12;31(4). pii: e00020-18. | CrossRef | PubMed |
Lakhundi S, Zhang K. Methicillin-Resistant Staphylococcus aureus: Molecular Characterization, Evolution, and Epidemiology. Clin Microbiol Rev. 2018 Sep 12;31(4). pii: e00020-18. | CrossRef | PubMed | Giudice Barbagelata MG, Pardo Casaretto L, Mota Ciganda MI, Gutiérrez Correa C, Algorta Rusiñol G, Varela Pensado G. Staphylococcus aureus portador del gen mecA sensible a oxacilina (OS-MRSA): otro desafío para los laboratorios de microbiología. Rev MEDICA DEL URUGUAY. 2018;34(4):240-3. | CrossRef |
Giudice Barbagelata MG, Pardo Casaretto L, Mota Ciganda MI, Gutiérrez Correa C, Algorta Rusiñol G, Varela Pensado G. Staphylococcus aureus portador del gen mecA sensible a oxacilina (OS-MRSA): otro desafío para los laboratorios de microbiología. Rev MEDICA DEL URUGUAY. 2018;34(4):240-3. | CrossRef | Bassetti M, Carnelutti A, Righi E. The role of methicillin-resistant Staphylococcus aureus in skin and soft tissue infections. Curr Opin Infect Dis. 2017;30(2):150-7. | CrossRef | PubMed |
Bassetti M, Carnelutti A, Righi E. The role of methicillin-resistant Staphylococcus aureus in skin and soft tissue infections. Curr Opin Infect Dis. 2017;30(2):150-7. | CrossRef | PubMed | Frost I, Van Boeckel TP, Pires J, Craig J, Laxminarayan R. Global Geographic Trends in Antimicrobial Resistance: The Role of International Travel. J Travel Med. 2019 May 22. pii: taz036. | CrossRef | PubMed |
Frost I, Van Boeckel TP, Pires J, Craig J, Laxminarayan R. Global Geographic Trends in Antimicrobial Resistance: The Role of International Travel. J Travel Med. 2019 May 22. pii: taz036. | CrossRef | PubMed | Seas C, Garcia C, Salles MJ, Labarca J, Luna C, Alvarez-Moreno C, et al. Staphylococcus aureus bloodstream infections in Latin America: Results of a multinational prospective cohort study. J Antimicrob Chemother. 2018;73(1):212-22. | CrossRef |
Seas C, Garcia C, Salles MJ, Labarca J, Luna C, Alvarez-Moreno C, et al. Staphylococcus aureus bloodstream infections in Latin America: Results of a multinational prospective cohort study. J Antimicrob Chemother. 2018;73(1):212-22. | CrossRef | Biedenbach DJ, Alm RA, Lahiri SD, Reiszner E, Hoban DJ, Sahm DF, et al. In Vitro Activity of Ceftaroline against Staphylococcus aureus Isolated in 2012 from Asia-Pacific Countries as Part of the AWARE Surveillance Program. Antimicrob Agents Chemother. 2015 Oct 26;60(1):343-7. | CrossRef | PubMed |
Biedenbach DJ, Alm RA, Lahiri SD, Reiszner E, Hoban DJ, Sahm DF, et al. In Vitro Activity of Ceftaroline against Staphylococcus aureus Isolated in 2012 from Asia-Pacific Countries as Part of the AWARE Surveillance Program. Antimicrob Agents Chemother. 2015 Oct 26;60(1):343-7. | CrossRef | PubMed | Planet PJ, Diaz L, Rios R, Arias CA. Global Spread of the Community-Associated Methicillin-Resistant Staphylococcus aureus USA300 Latin American Variant. J Infect Dis. 2016 Nov 15;214(10):1609-1610. | PubMed |
Planet PJ, Diaz L, Rios R, Arias CA. Global Spread of the Community-Associated Methicillin-Resistant Staphylococcus aureus USA300 Latin American Variant. J Infect Dis. 2016 Nov 15;214(10):1609-1610. | PubMed | Duplessis C, Crum-Cianflone NF. Ceftaroline: A New Cephalosporin with Activity against Methicillin-Resistant Staphylococcus aureus (MRSA). Clin Med Rev Ther. 2011 Feb 10;3. pii: a2466. | PubMed |
Duplessis C, Crum-Cianflone NF. Ceftaroline: A New Cephalosporin with Activity against Methicillin-Resistant Staphylococcus aureus (MRSA). Clin Med Rev Ther. 2011 Feb 10;3. pii: a2466. | PubMed | Hoban D, Biedenbach D, Sahm D, Reiszner E, Iaconis J. Activity of ceftaroline and comparators against pathogens isolated from skin and soft tissue infections in Latin America - results of AWARE surveillance 2012. Braz J Infect Dis. 2015 Nov-Dec;19(6):596-603. | CrossRef | PubMed |
Hoban D, Biedenbach D, Sahm D, Reiszner E, Iaconis J. Activity of ceftaroline and comparators against pathogens isolated from skin and soft tissue infections in Latin America - results of AWARE surveillance 2012. Braz J Infect Dis. 2015 Nov-Dec;19(6):596-603. | CrossRef | PubMed | Choo EJ, Chambers HF. Treatment of Methicillin-Resistant Staphylococcus aureus Bacteremia. Infect Chemother. 2016 Dec;48(4):267-273. | CrossRef | PubMed |
Choo EJ, Chambers HF. Treatment of Methicillin-Resistant Staphylococcus aureus Bacteremia. Infect Chemother. 2016 Dec;48(4):267-273. | CrossRef | PubMed | Sader HS, Castanheira M, Farrell DJ, Flamm RK, Mendes RE, Jones RN. Tigecycline antimicrobial activity tested against clinical bacteria from Latin American medical centres: results from SENTRY Antimicrobial Surveillance Program (2011-2014). Int J Antimicrob Agents. 2016 Aug;48(2):144-50. | CrossRef | PubMed |
Sader HS, Castanheira M, Farrell DJ, Flamm RK, Mendes RE, Jones RN. Tigecycline antimicrobial activity tested against clinical bacteria from Latin American medical centres: results from SENTRY Antimicrobial Surveillance Program (2011-2014). Int J Antimicrob Agents. 2016 Aug;48(2):144-50. | CrossRef | PubMed | Vega S, Dowzicky MJ. Antimicrobial susceptibility among Gram-positive and Gram-negative organisms collected from the Latin American region between 2004 and 2015 as part of the Tigecycline Evaluation and Surveillance Trial. Ann Clin Microbiol Antimicrob. 2017;16(1):1–16. | CrossRef |
Vega S, Dowzicky MJ. Antimicrobial susceptibility among Gram-positive and Gram-negative organisms collected from the Latin American region between 2004 and 2015 as part of the Tigecycline Evaluation and Surveillance Trial. Ann Clin Microbiol Antimicrob. 2017;16(1):1–16. | CrossRef | Damodaran SE, Madhan S. Telavancin: A novel lipoglycopeptide antibiotic. J Pharmacol Pharmacother. 2011 Apr;2(2):135-7. | CrossRef | PubMed |
Damodaran SE, Madhan S. Telavancin: A novel lipoglycopeptide antibiotic. J Pharmacol Pharmacother. 2011 Apr;2(2):135-7. | CrossRef | PubMed | Mendes RE, Sader HS, Smart JI, Castanheira M, Flamm RK. Update of the activity of telavancin against a global collection of Staphylococcus aureus causing bacteremia, including endocarditis (2011-2014). Eur J Clin Microbiol Infect Dis. 2017 Jun;36(6):1013-1017. | CrossRef | PubMed |
Mendes RE, Sader HS, Smart JI, Castanheira M, Flamm RK. Update of the activity of telavancin against a global collection of Staphylococcus aureus causing bacteremia, including endocarditis (2011-2014). Eur J Clin Microbiol Infect Dis. 2017 Jun;36(6):1013-1017. | CrossRef | PubMed | Hulten KG. The changing epidemiology of pneumococcal diseases. Lancet Infect Dis. 2018 Sep;18(9):929-930. | CrossRef | PubMed |
Hulten KG. The changing epidemiology of pneumococcal diseases. Lancet Infect Dis. 2018 Sep;18(9):929-930. | CrossRef | PubMed | Cillóniz C, Garcia-Vidal C, Ceccato A, Torres A. Antimicrobial Resistance Among Streptococcus pneumoniae. En: Antimicrobial Resistance in the 21st Century. Second. Gewerbestrasse: Springer; 2018:13–39. | Link |
Cillóniz C, Garcia-Vidal C, Ceccato A, Torres A. Antimicrobial Resistance Among Streptococcus pneumoniae. En: Antimicrobial Resistance in the 21st Century. Second. Gewerbestrasse: Springer; 2018:13–39. | Link | Agudelo CI, DeAntonio R, Castañeda E. Authors reply "Streptococcus pneumoniae serotype 19A in Latin America and the Caribbean 2010-2015: A systematic review and a time series analysis". Vaccine. 2019 Jun 6;37(26):3387. | CrossRef | PubMed |
Agudelo CI, DeAntonio R, Castañeda E. Authors reply "Streptococcus pneumoniae serotype 19A in Latin America and the Caribbean 2010-2015: A systematic review and a time series analysis". Vaccine. 2019 Jun 6;37(26):3387. | CrossRef | PubMed | Hawkins P, Mercado E, Chochua S, Castillo ME, Reyes I, Chaparro E, et al. Key features of invasive pneumococcal isolates recovered in Lima, Peru determined through whole genome sequencing. Int J Med Microbiol. 2017 Oct;307(7):415-421. | CrossRef | PubMed |
Hawkins P, Mercado E, Chochua S, Castillo ME, Reyes I, Chaparro E, et al. Key features of invasive pneumococcal isolates recovered in Lima, Peru determined through whole genome sequencing. Int J Med Microbiol. 2017 Oct;307(7):415-421. | CrossRef | PubMed | Mulani MS, Kamble EE, Kumkar SN, Tawre MS, Pardesi KR. Emerging Strategies to Combat ESKAPE Pathogens in the Era of Antimicrobial Resistance: A Review. Front Microbiol. 2019 Apr 1;10:539. | CrossRef | PubMed |
Mulani MS, Kamble EE, Kumkar SN, Tawre MS, Pardesi KR. Emerging Strategies to Combat ESKAPE Pathogens in the Era of Antimicrobial Resistance: A Review. Front Microbiol. 2019 Apr 1;10:539. | CrossRef | PubMed | Guzmán-Blanco M, Labarca JA, Villegas MV, Gotuzzo E; Latin America Working Group on Bacterial Resistance. Extended spectrum β-lactamase producers among nosocomial Enterobacteriaceae in Latin America. Braz J Infect Dis. 2014 Jul-Aug;18(4):421-33. | CrossRef | PubMed |
Guzmán-Blanco M, Labarca JA, Villegas MV, Gotuzzo E; Latin America Working Group on Bacterial Resistance. Extended spectrum β-lactamase producers among nosocomial Enterobacteriaceae in Latin America. Braz J Infect Dis. 2014 Jul-Aug;18(4):421-33. | CrossRef | PubMed | Kehl SC, Dowzicky MJ. Global assessment of antimicrobial susceptibility among Gram-negative organisms collected from pediatric patients between 2004 and 2012: results from the Tigecycline Evaluation and Surveillance Trial. J Clin Microbiol. 2015 Apr;53(4):1286-93. | CrossRef | PubMed |
Kehl SC, Dowzicky MJ. Global assessment of antimicrobial susceptibility among Gram-negative organisms collected from pediatric patients between 2004 and 2012: results from the Tigecycline Evaluation and Surveillance Trial. J Clin Microbiol. 2015 Apr;53(4):1286-93. | CrossRef | PubMed | Jiménez A, Alvarado A, Gómez F, Carrero G, Fajardo C. [Risk factors associated with the isolation of extended spectrum betalactamases producing Escherichia coli or Klebsiella pneumoniae in a tertiary care hospital in Colombia]. Biomedica. 2014 Apr;34 Suppl 1:16-22. | CrossRef | PubMed |
Jiménez A, Alvarado A, Gómez F, Carrero G, Fajardo C. [Risk factors associated with the isolation of extended spectrum betalactamases producing Escherichia coli or Klebsiella pneumoniae in a tertiary care hospital in Colombia]. Biomedica. 2014 Apr;34 Suppl 1:16-22. | CrossRef | PubMed | Pereira A, Fariña N, de Vega M, González P, Rodríguez F, de Figueredo L, et al. Enterobacterias productoras de Betalactamasas de espectro extendido aisladas de pacientes ambulatorios y hospitalizados en un Laboratorio privado de Asunción Extended-spectrum-Betalactamases producing enterobacteriaceae isolated from outpatient and hospit. Investig Cienc Salud Investig Cienc Salud. 2016;1414(11):17-2417.
| CrossRef |
Pereira A, Fariña N, de Vega M, González P, Rodríguez F, de Figueredo L, et al. Enterobacterias productoras de Betalactamasas de espectro extendido aisladas de pacientes ambulatorios y hospitalizados en un Laboratorio privado de Asunción Extended-spectrum-Betalactamases producing enterobacteriaceae isolated from outpatient and hospit. Investig Cienc Salud Investig Cienc Salud. 2016;1414(11):17-2417.
| CrossRef | Kazmierczak KM, Lob SH, Hoban DJ, Hackel MA, Badal RE, Bouchillon SK. Characterization of extended-spectrum beta-lactamases and antimicrobial resistance of Klebsiella pneumoniae in intra-abdominal infection isolates in Latin America, 2008-2012. Results of the Study for Monitoring Antimicrobial Resistance Trends. Diagn Microbiol Infect Dis. 2015 Jul;82(3):209-14. | CrossRef | PubMed |
Kazmierczak KM, Lob SH, Hoban DJ, Hackel MA, Badal RE, Bouchillon SK. Characterization of extended-spectrum beta-lactamases and antimicrobial resistance of Klebsiella pneumoniae in intra-abdominal infection isolates in Latin America, 2008-2012. Results of the Study for Monitoring Antimicrobial Resistance Trends. Diagn Microbiol Infect Dis. 2015 Jul;82(3):209-14. | CrossRef | PubMed | Dropa M, Balsalobre LC, Lincopan N, Matté GR, Matté MH. Complex class 1 integrons harboring CTX-M-2-encoding genes in clinical Enterobacteriaceae from a hospital in Brazil. J Infect Dev Ctries. 2015 Aug 29;9(8):890-7. | CrossRef | PubMed |
Dropa M, Balsalobre LC, Lincopan N, Matté GR, Matté MH. Complex class 1 integrons harboring CTX-M-2-encoding genes in clinical Enterobacteriaceae from a hospital in Brazil. J Infect Dev Ctries. 2015 Aug 29;9(8):890-7. | CrossRef | PubMed | Karlowsky JA, Hoban DJ, Hackel MA, Lob SH, Sahm DF. Resistance among Gram-negative ESKAPE pathogens isolated from hospitalized patients with intra-abdominal and urinary tract infections in Latin American countries: SMART 2013-2015. Braz J Infect Dis. 2017 May - Jun;21(3):343-348. | CrossRef | PubMed |
Karlowsky JA, Hoban DJ, Hackel MA, Lob SH, Sahm DF. Resistance among Gram-negative ESKAPE pathogens isolated from hospitalized patients with intra-abdominal and urinary tract infections in Latin American countries: SMART 2013-2015. Braz J Infect Dis. 2017 May - Jun;21(3):343-348. | CrossRef | PubMed | Furlan JPR, Stehling EG. Detection of β-lactamase encoding genes in feces, soil and water from a Brazilian pig farm. Environ Monit Assess. 2018 Jan 10;190(2):76. | CrossRef | PubMed |
Furlan JPR, Stehling EG. Detection of β-lactamase encoding genes in feces, soil and water from a Brazilian pig farm. Environ Monit Assess. 2018 Jan 10;190(2):76. | CrossRef | PubMed | Wong D, Nielsen TB, Bonomo RA, Pantapalangkoor P, Luna B, Spellberg B. Clinical and Pathophysiological Overview of Acinetobacter Infections: a Century of Challenges. Clin Microbiol Rev. 2017 Jan;30(1):409-447. | CrossRef | PubMed |
Wong D, Nielsen TB, Bonomo RA, Pantapalangkoor P, Luna B, Spellberg B. Clinical and Pathophysiological Overview of Acinetobacter Infections: a Century of Challenges. Clin Microbiol Rev. 2017 Jan;30(1):409-447. | CrossRef | PubMed | Levy-Blitchtein S, Roca I, Plasencia-Rebata S, Vicente-Taboada W, Velásquez-Pomar J, Muñoz L, et al. Emergence and spread of carbapenem-resistant Acinetobacter baumannii international clones II and III in Lima, Peru. Emerg Microbes Infect. 2018 Jul 4;7(1):119. | CrossRef | PubMed |
Levy-Blitchtein S, Roca I, Plasencia-Rebata S, Vicente-Taboada W, Velásquez-Pomar J, Muñoz L, et al. Emergence and spread of carbapenem-resistant Acinetobacter baumannii international clones II and III in Lima, Peru. Emerg Microbes Infect. 2018 Jul 4;7(1):119. | CrossRef | PubMed | Gonzalez-Villoria AM, Valverde-Garduno V. Antibiotic-Resistant Acinetobacter baumannii Increasing Success Remains a Challenge as a Nosocomial Pathogen. J Pathog. 2016;2016:7318075. | CrossRef | PubMed |
Gonzalez-Villoria AM, Valverde-Garduno V. Antibiotic-Resistant Acinetobacter baumannii Increasing Success Remains a Challenge as a Nosocomial Pathogen. J Pathog. 2016;2016:7318075. | CrossRef | PubMed | Logan LK, Medernach RL, Rispens JR, Marshall SH, Hujer AM, Domitrovic TN, et al. Community Origins and Regional Differences Highlight Risk of Plasmid-mediated Fluoroquinolone Resistant Enterobacteriaceae Infections in Children. Pediatr Infect Dis J. 2019 Jun;38(6):595-599.
| CrossRef | PubMed |
Logan LK, Medernach RL, Rispens JR, Marshall SH, Hujer AM, Domitrovic TN, et al. Community Origins and Regional Differences Highlight Risk of Plasmid-mediated Fluoroquinolone Resistant Enterobacteriaceae Infections in Children. Pediatr Infect Dis J. 2019 Jun;38(6):595-599.
| CrossRef | PubMed | Kazmierczak KM, Biedenbach DJ, Hackel M, Rabine S, de Jonge BL, Bouchillon SK, et al. Global Dissemination of blaKPC into Bacterial Species beyond Klebsiella pneumoniae and In Vitro Susceptibility to Ceftazidime-Avibactam and Aztreonam-Avibactam. Antimicrob Agents Chemother. 2016 Jul 22;60(8):4490-500. | CrossRef | PubMed |
Kazmierczak KM, Biedenbach DJ, Hackel M, Rabine S, de Jonge BL, Bouchillon SK, et al. Global Dissemination of blaKPC into Bacterial Species beyond Klebsiella pneumoniae and In Vitro Susceptibility to Ceftazidime-Avibactam and Aztreonam-Avibactam. Antimicrob Agents Chemother. 2016 Jul 22;60(8):4490-500. | CrossRef | PubMed | Bado I, Papa-Ezdra R, Delgado-Blas JF, Gaudio M, Gutiérrez C, Cordeiro NF, et al. Molecular Characterization of Carbapenem-Resistant Acinetobacter baumannii in the Intensive Care Unit of Uruguay's University Hospital Identifies the First rmtC Gene in the Species. Microb Drug Resist. 2018 Sep;24(7):1012-1019. | CrossRef | PubMed |
Bado I, Papa-Ezdra R, Delgado-Blas JF, Gaudio M, Gutiérrez C, Cordeiro NF, et al. Molecular Characterization of Carbapenem-Resistant Acinetobacter baumannii in the Intensive Care Unit of Uruguay's University Hospital Identifies the First rmtC Gene in the Species. Microb Drug Resist. 2018 Sep;24(7):1012-1019. | CrossRef | PubMed | Marquez-Ortiz RA, Haggerty L, Olarte N, Duarte C, Garza-Ramos U, Silva-Sanchez J, et al. Genomic Epidemiology of NDM-1-Encoding Plasmids in Latin American Clinical Isolates Reveals Insights into the Evolution of Multidrug Resistance. Genome Biol Evol. 2017 Jun 1;9(6):1725-1741. | CrossRef | PubMed |
Marquez-Ortiz RA, Haggerty L, Olarte N, Duarte C, Garza-Ramos U, Silva-Sanchez J, et al. Genomic Epidemiology of NDM-1-Encoding Plasmids in Latin American Clinical Isolates Reveals Insights into the Evolution of Multidrug Resistance. Genome Biol Evol. 2017 Jun 1;9(6):1725-1741. | CrossRef | PubMed | Jones-Dias D, Manageiro V, Caniça M. Influence of agricultural practice on mobile bla genes: IncI1-bearing CTX-M, SHV, CMY and TEM in Escherichia coli from intensive farming soils. Environ Microbiol. 2016 Jan;18(1):260-72. | CrossRef | PubMed |
Jones-Dias D, Manageiro V, Caniça M. Influence of agricultural practice on mobile bla genes: IncI1-bearing CTX-M, SHV, CMY and TEM in Escherichia coli from intensive farming soils. Environ Microbiol. 2016 Jan;18(1):260-72. | CrossRef | PubMed | Furlan JPR, Stehling EG. Detection of β-lactamase encoding genes in feces, soil and water from a Brazilian pig farm. Environ Monit Assess. 2018 Jan 10;190(2):76. | CrossRef | PubMed |
Furlan JPR, Stehling EG. Detection of β-lactamase encoding genes in feces, soil and water from a Brazilian pig farm. Environ Monit Assess. 2018 Jan 10;190(2):76. | CrossRef | PubMed | Pitondo-Silva A, Devechio BB, Moretto JA, Stehling EG. High prevalence of bla(VIM-1) gene in bacteria from Brazilian soil. Can J Microbiol. 2016 Oct;62(10):820-826. | PubMed |
Pitondo-Silva A, Devechio BB, Moretto JA, Stehling EG. High prevalence of bla(VIM-1) gene in bacteria from Brazilian soil. Can J Microbiol. 2016 Oct;62(10):820-826. | PubMed | Braykov NP, Eisenberg JN, Grossman M, Zhang L, Vasco K, Cevallos W, et al. Antibiotic Resistance in Animal and Environmental Samples Associated with Small-Scale Poultry Farming in Northwestern Ecuador. mSphere. 2016 Feb 10;1(1). pii: e00021-15. | CrossRef | PubMed |
Braykov NP, Eisenberg JN, Grossman M, Zhang L, Vasco K, Cevallos W, et al. Antibiotic Resistance in Animal and Environmental Samples Associated with Small-Scale Poultry Farming in Northwestern Ecuador. mSphere. 2016 Feb 10;1(1). pii: e00021-15. | CrossRef | PubMed | Guo X, Stedtfeld RD, Hedman H, Eisenberg JNS, Trueba G, Yin D, et al. Antibiotic Resistome Associated with Small-Scale Poultry Production in Rural Ecuador. Environ Sci Technol. 2018 Aug 7;52(15):8165-8172. | CrossRef | PubMed |
Guo X, Stedtfeld RD, Hedman H, Eisenberg JNS, Trueba G, Yin D, et al. Antibiotic Resistome Associated with Small-Scale Poultry Production in Rural Ecuador. Environ Sci Technol. 2018 Aug 7;52(15):8165-8172. | CrossRef | PubMed | Moser KA, Zhang L, Spicknall I, Braykov NP, Levy K, Marrs CF, et al. The Role of Mobile Genetic Elements in the Spread of Antimicrobial-Resistant Escherichia coli From Chickens to Humans in Small-Scale Production Poultry Operations in Rural Ecuador. Am J Epidemiol. 2018 Mar 1;187(3):558-567. | CrossRef | PubMed |
Moser KA, Zhang L, Spicknall I, Braykov NP, Levy K, Marrs CF, et al. The Role of Mobile Genetic Elements in the Spread of Antimicrobial-Resistant Escherichia coli From Chickens to Humans in Small-Scale Production Poultry Operations in Rural Ecuador. Am J Epidemiol. 2018 Mar 1;187(3):558-567. | CrossRef | PubMed | Hedman HD, Eisenberg JNS, Vasco KA, Blair CN, Trueba G, Berrocal VJ, et al. High Prevalence of Extended-Spectrum Beta-Lactamase CTX-M-Producing Escherichia coli in Small-Scale Poultry Farming in Rural Ecuador. Am J Trop Med Hyg. 2019 Feb;100(2):374-376. | CrossRef | PubMed |
Hedman HD, Eisenberg JNS, Vasco KA, Blair CN, Trueba G, Berrocal VJ, et al. High Prevalence of Extended-Spectrum Beta-Lactamase CTX-M-Producing Escherichia coli in Small-Scale Poultry Farming in Rural Ecuador. Am J Trop Med Hyg. 2019 Feb;100(2):374-376. | CrossRef | PubMed | Vinueza-Burgos C, Baquero M, Medina J, De Zutter L. Occurrence, genotypes and antimicrobial susceptibility of Salmonella collected from the broiler production chain within an integrated poultry company. Int J Food Microbiol. 2019 Jun 16;299:1-7. | CrossRef | PubMed |
Vinueza-Burgos C, Baquero M, Medina J, De Zutter L. Occurrence, genotypes and antimicrobial susceptibility of Salmonella collected from the broiler production chain within an integrated poultry company. Int J Food Microbiol. 2019 Jun 16;299:1-7. | CrossRef | PubMed | Maciel MJ, Machado G, Avancini CAM. Investigation of resistance of Salmonella spp. isolated from products and raw material of animal origin (swine and poultry)to antibiotics and disinfectants. Rev Bras Saúde Prod Anim. 2019;20:1-13. | CrossRef |
Maciel MJ, Machado G, Avancini CAM. Investigation of resistance of Salmonella spp. isolated from products and raw material of animal origin (swine and poultry)to antibiotics and disinfectants. Rev Bras Saúde Prod Anim. 2019;20:1-13. | CrossRef | Huamán-Chacón LE, Edgar G-E. Escherichia coli Productor de betalactamasas de espectro extendido en pollos para consumo humano. Rev Peru Med Exp Salud Publica. 2019;36(2):361-2. | CrossRef |
Huamán-Chacón LE, Edgar G-E. Escherichia coli Productor de betalactamasas de espectro extendido en pollos para consumo humano. Rev Peru Med Exp Salud Publica. 2019;36(2):361-2. | CrossRef | Carvajal B. E, Hernández A. W, Torres C. M, López V. D, Rueda G. E, Vásquez de Díaz MC. Resistencia antimicrobiana de cepas de Escherichia coli aisladas de contenidos de bursa de Fabricio de aves para engorde. Rev Investig Vet del Perú. 2019;30(1):430-7. | CrossRef |
Carvajal B. E, Hernández A. W, Torres C. M, López V. D, Rueda G. E, Vásquez de Díaz MC. Resistencia antimicrobiana de cepas de Escherichia coli aisladas de contenidos de bursa de Fabricio de aves para engorde. Rev Investig Vet del Perú. 2019;30(1):430-7. | CrossRef | Ferreira JC, Penha Filho RAC, Andrade LN, Berchieri Junior A, Darini ALC. Diversity of plasmids harboring bla(CMY-2) in multidrug-resistant Escherichia coli isolated from poultry in Brazil. Diagn Microbiol Infect Dis. 2017 Aug;88(4):361-364. | CrossRef | PubMed |
Ferreira JC, Penha Filho RAC, Andrade LN, Berchieri Junior A, Darini ALC. Diversity of plasmids harboring bla(CMY-2) in multidrug-resistant Escherichia coli isolated from poultry in Brazil. Diagn Microbiol Infect Dis. 2017 Aug;88(4):361-364. | CrossRef | PubMed |