La publicación de estas Actas Científicas ha sido posible gracias a una colaboración editorial entre Medwave y la Sociedad Chilena de Nefrología.
Se presentan los resultados de los estudios llevados a cabo en los últimos 18 meses, con el objeto de buscar polimorfismos e indagar en las bases genéticas de las complicaciones asociadas con diabetes mellitus, específicamente la nefropatía diabética. La investigación se está realizando con el apoyo de un proyecto FONIS, que es un convenio entre el Ministerio de Salud y CONICYT, y un proyecto multidisciplinario de la Universidad de Chile y en ella participaron la Unidad de Diabetes de la Facultad de Medicina, Sede Occidente de la Universidad de Chile, el Laboratorio de Bioquímica del Departamento de Bioquímica y Biología Molecular de la Facultad de Ciencias Químicas y Farmacéuticas de la Universidad de Chile, la Sección de Nefrología y el Centro Cardiovascular del Hospital Clínico de la Universidad de Chile.
Se sabe que, en condiciones clínico-metabólicas similares, algunas personas padecen nefropatía diabética y otras no, lo que se explica porque existe un factor genético, una susceptibilidad que predispone a algunas personas, portadoras del cuadro de diabetes, a mayor riesgo de desarrollar nefropatía. Los genes causantes de este fenómeno pueden ser varios, por lo que es necesario acotar el campo de estudio. También se sabe que el mal control metabólico es una condición necesaria, aunque no suficiente, para presentar nefropatía, la que se debe a un fenómeno multifactorial. Sólo en un porcentaje de los diabéticos se desarrolla nefropatía diabética y un porcentaje variable de los nefrópatas evolucionan a insuficiencia renal terminal. Para estudiar esta particularidad decidimos analizar los polimorfismos en un gen escogido, en este caso el de la nefrina, por su importancia en la función de filtración del glomérulo. El objetivo era determinar si estos polimorfismos genéticos podrían estar asociados con un cuadro de mayor susceptibilidad de nefropatía diabética.
Se incluyó en el estudio un total de 222 hombres y mujeres de 18 a 74 años de edad, de los cuales 60 eran controles (40 personas sanas y 20 nefrópatas no diabéticos) (Fig. 1). Se formaron grupos de diabéticos tipo 1 y tipo 2, y ambos se subdividieron según el grado de compromiso renal en normo, micro y macroalbuminúricos.
Figura 1. Diseño del estudio: características de los grupos
A cada persona se le extrajo una muestra de sangre periférica, previa firma de una carta de consentimiento; ambos proyectos fueron aprobados por los respectivos comités de ética. Los distintos grupos se sometieron a exámenes clínicos (sexo, edad, presión arterial e índice de masa corporal) y bioquímicos (glicemia, creatinina, Hb1Ac, niveles lipídicos y albuminuria), y de las mismas muestras que se tomaron para los exámenes bioquímicos se extrajo ADN para efectuar el análisis de la presencia de polimorfismos en nefrina, mediante la técnica SSCP-PCR (Single Strand Conformational Polymorphism).
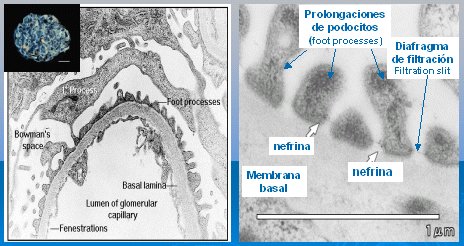
El interés en la nefrina, que fue descubierta en el año 1998, se debe a que se considera como la causante del síndrome nefrótico congénito tipo finlandés. La mutación del gen que codifica para esta proteína origina proteinuria masiva in utero y ocasiona la muerte de estos lactantes a los dos o tres meses de vida. Por lo tanto, esta proteína tiene una participación importante en la función renal. En un glomérulo en el que se destacan unas células diferenciadas (Fig. 2) que son los podocitos, cuyas prolongaciones están adosadas a la lámina basal; entre las diversas prolongaciones o foot processes de los podocitos del glomérulo hay una barrera funcional, que es la membrana de filtración; en ese lugar se ubica la nefrina, que se detecta mediante técnicas inmunohistoquímicas como pequeños puntos entre las uniones de las prolongaciones de podocitos adyacentes.
Figura 2. Localización anatómica de la nefrina en el glomérulo
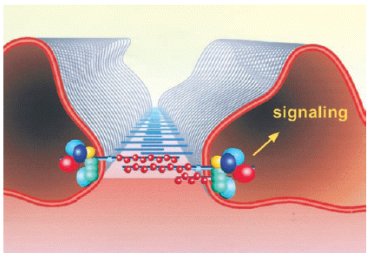
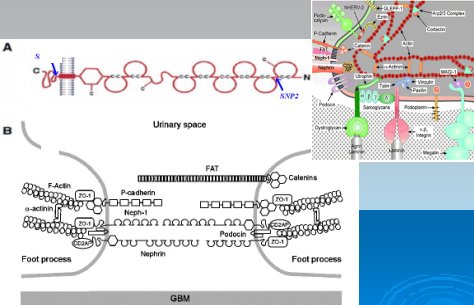
La función de la nefrina es estructural: es una proteína grande que pertenece a la superfamilia de las inmunoglobulinas y cada una de las pequeñas esferas rojas (Fig. 3) representa dominio de inmunoglobulina. Entre una prolongación de podocito y la prolongación vecina aparece una estructura de interdigitación de proteínas, la que ocupa un espacio de 40 nanómetros, aproximadamente, y constituye la membrana de filtración. La nefrina, en su porción extracelular, cumple una función estructural importante, ya que define el paso de moléculas. Tiene un dominio de fibronectina, un dominio transmembrana y, hacia el interior de la célula, se conecta con una serie de proteínas y cumple una función intracelular de señalización. En el esquema se ve la nefrina como una proteína de gran tamaño, pero no es la única que proyecta sus dominios extracelulares hacia el espacio extracelular; otras proteínas que incluso son de la misma familia, como la Neph-1 y la podocina, forman dímeros entre ellas, homodímeros o heterodímeros de nefrina, como con Neph-1, etc.
Hacia el interior de la célula hay conexiones muy importantes: por ejemplo, con podocina, que también es una proteína que forma parte de la membrana de filtración, y con otras, como CD2AP, con la cual se comunica a nivel intracelular y se conecta hacia el interior de la célula en una cascada de transcripción de señales. Esta cascada de señalización a través de kinasas, es importante para la sobrevida del podocito. Se ha visto que no sólo en el síndrome nefrótico hay proteínas truncadas que alteran esta estructura; también en enfermedades renales proteinúricas adquiridas hay, probablemente, una alteración en la conexión intracelular con esas otras proteínas, de modo que se pierden las conexiones con la lámina basal y ocurren alteraciones con la señalización, las que luego causan apoptosis celular y pérdida de podocitos funcionales. En consecuencia, esta nefrina es una proteína importante desde ambos puntos de vista, tanto estructural como en la señalización intracelular (Fig. 4).
Figura 3. Función estructural y de transducción de señales de nefrina en el podocito (J Am Soc Nephrol 15: 1382-1391, 2004
Figura 4. Función de la nefrina en el podocito
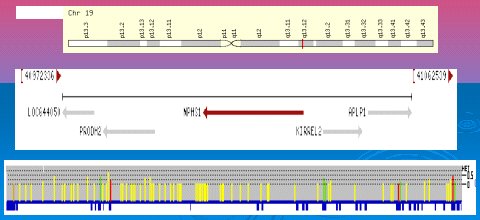
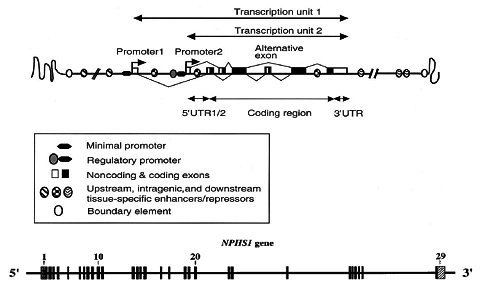
En 1999 se publicó que el gen que codifica para la nefrina está ubicado en el cromosoma 19, en la región particular 19q13.1, es decir, en el brazo largo. Se observa que en el vecindario de este gen, que se denomina NPHS1, aparecen otros genes que también están relacionados con la función renal, como el gen Kirrel2, que codifica para filtrina, una proteína similar a nefrina. El gen de nefrina, cuya estructura se muestra en forma muy esquemática (Fig. 5), es bastante grande; está formado por 29 exones y cubre varios kilobases de ADN. La estructura modular de exones e intrones es típica de los genes eucarióticos; sólo los exones participan por ultimo en lo que será la proteína propiamente tal.
Aparte de las mutaciones que puede sufrir un gen y que van a dar un resultado patológico o patogénico, se encuentra una serie de variaciones normales entre individuos de una población. En humanos se han descrito ya 82 polimorfismos genéticos del gen de nefrina humana, que serían normales y se ilustran en las barras amarillas (Fig. 5). Las barras en amarillo y hacia arriba representan polimorfismos normales dentro de los intrones, es decir, dentro de aquellas zonas que no son codificantes de un gen; las verdes, que son menos, son polimorfismos que están en exones, es decir, dentro de las zonas que van a dar lugar a la proteína, pero no originan un cambio de aminoácidos; y en rojo hay tres polimorfismos descritos que llevan a un cambio en el aminoácido en la proteína: son los llamados polimorfismos no sinónimos, que suelen ser los más interesantes, porque dentro de la condición de normalidad podrían conducir a un cambio, una modulación, alguna sutileza en cuanto a su interacción con algunas proteínas.
Figura 5. Localización cromosomal y estructura del gen de la nefrina humana. Cromosoma: 19; región: 19q13. 151,847 bases. Orientación: minus strand
Un esquema general ilustra la complejidad de un gen prototipo eucariótico, entre los cuales estamos los seres humanos (Fig. 6). Cada gen se puede componer de múltiples exones e intrones; estos exones e intrones, se pueden procesar en forma diferencial en distintos tejidos, de modo que lo que es exón en un tejido, puede ser intrón en otro y viceversa. En el caso del gen de nefrina, está formado por 29 módulos y presenta distintos transcritos, es decir, sufre distintos procesamientos en distintos tejidos. Se sabe que no sólo se expresa en riñón, sino también en páncreas y también, por alguna razón que aún no se conoce, se expresa en cerebelo.
Figura 6. Esquema teórico de un gen eucariótico
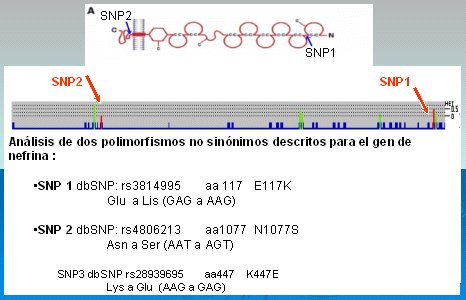
El objetivo del estudio era determinar, en primer lugar, si hay alguna asociación o alguna diferencia en la frecuencia de estos polimorfismos en nuestras poblaciones de diabéticos. En una comunicación escandinava se había descrito que no había asociación entre estos polimorfismos no sinónimos y la frecuencia de nefropatía diabética, pero esto podría variar de una población a otra. Se estudió una región que comprende el polimorfismo SNP1 dbSNP: rs3814995 y otra región que comprende un segundo polimorfismo, el SNP2 dbSNP: rs4806213. El polimorfismo que se denominó 1, para simplificar, se encuentra en la parte extracelular y podría ser que un cambio en él modulara o alterara la interacción con otras proteínas de la membrana del diafragma de filtración. El otro polimorfismo se encuentra justo en el lado interior del dominio transmembrana, de modo que su estudio también podría ser interesante. El polimorfismo 1 codifica para un cambio de glutámico a lisina, un cambio puntual de una sola base y podría ser interesante, porque significa un cambio de carga de un aminoácido. El segundo polimorfismo causa un cambio de asparragina por serina, también es un cambio puntual y codifica para la aparición de una serina en la posición 1077, o sea, en la región carboxilo-terminal de la proteína (Fig. 7).
Figura 7. Regiones del gen escogidas para el análisis de polimorfismos
Para realizar estos estudios, lo primero que se hizo, después de obtener las muestras de sangre, fue extraer el ADN mediante el sistema comercial QIAamp DNA blood mini kit, que es eficaz, de buen rendimiento y reproducible, y permite obtener ADN a partir de 200 microlitros de sangre. Se aplicó un sistema de barrido o tamizaje para la detección de cambios puntuales en la secuencia. Para ello se amplifica un segmento pequeño de ADN, de no más de 300 pares de bases. Posteriormente estos segmentos se separan mediante un sistema de electroforesis y se ajustan las condiciones, de tal manera que el cambio de una base produce un cambio de conformación de la hebra, las que se separan en condiciones no desnaturantes, de modo que se visualiza este cambio.
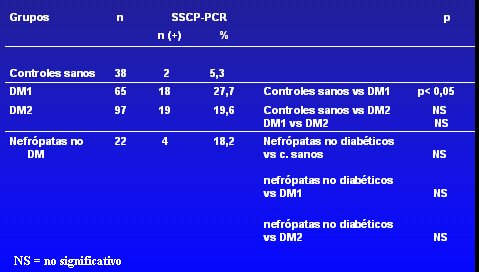
El sistema es tan sensible que el cambio de una base debiera producir un cambio de conformación, que se ve como un cambio de migración de esa banda en el gel. En la Tabla I. se muestra el resultado de la frecuencia de la presencia de los polimorfismos que se analizaron en todos los pacientes. En el caso del polimorfismo de la región 2, si no aparece el polimorfismo, se observa un cierto patrón de bandas; en cambio, cuando existe un polimorfismo se produce un cambio claro en el patrón de migración, de modo que desaparece una banda. Lo anterior se observó en pacientes diabéticos tipo 1 y tipo 2.
Tabla I. Polimorfismo del gen NPHS1 en controles sanos, pacientes diabéticos tipo 1 y 2 y nefrópatas no diabéticos
El polimorfismo de la región denominada 2, es decir, el que corresponde a una variación en la región de la proteína cercana al dominio transmembrana, es poco frecuente en controles sanos y sólo se encontró en 5% de ellos; en cambio, 27% de los diabéticos tipo 1 presentaban este cambio, lo mismo que 19,6% de los diabéticos tipo 2 y 18,2% de los nefrópatas no diabéticos. Sólo fue estadísticamente significativa la diferencia entre controles sanos y diabéticos tipo 1. También se comprobó que no había diferencia significativa según grado de daño renal; es decir, no había diferencia entre los grupos normo, micro y macroalbuminúricos.
Es preciso reconocer que no basta con usar sólo una técnica de barrido, porque sólo se observa un cambio de migración, sino que además se debe verificar que el polimorfismo que se está buscando efectivamente corresponda a lo que se postula. Por eso se procedió a efectuar la secuenciación de ADN, para confirmar lo anterior. El análisis del polimorfismo en la región 1 indicaba que estaba presente en nuestra población, pero al hacer el análisis de la secuencia de la región 2 se descubrió que el polimorfismo que se había detectado no era el que se buscaba, sino que apareció dentro de la misma región un segundo polimorfismo, en el segmento que se había amplificado; o sea, no sólo estaba el polimorfismo que se buscaba, sino que además había una segunda variante en esta zona. Se trataba de un polimorfismo en un intrón vecino, dado que el segmento amplificado cubría parte de un exón y parte de un intrón. En la gran mayoría de los pacientes el polimorfismo no era el del exón, sino que el polimorfismo estaba en el intrón. Este polimorfismo intrónico en sí estaba descrito, y corresponde al polimorfismo SNP rs# 466452, pero no se había relacionado su presencia con nefropatía diabética ni con otra enfermedad, y este hecho resultaba inesperado y nuevo. Es probable que una variante en un intrón tenga menos consecuencias y afecte menos la función biológica que un cambio en lo que va a ser la proteína propiamente tal. Sin embargo, si bien las secuencias de los intrones no se reflejarán en las proteínas, es posible que participen en regular el procesamiento de los propios intrones y definan qué va a ser intrón y qué va ser exón; no son necesariamente zonas sin función.
El paso siguiente era determinar la función que podría cumplir este polimorfismo y el interés que podría tener, considerando que cerca de 90% de nuestros pacientes presentaban el polimorfismo en el intrón y sólo 5% a 8% de ellos tenían el polimorfismo en la proteína. Se contaba con encontrar un cambio en el SNP rs4806213, pero en la gran mayoría de los controles sanos de los distintos grupos había lo previsto, esto es, una adenina en esa posición, y sólo algunos presentaban una guanina; en cambio, muchos pacientes presentaron una citosina en la posición del polimorfismo intrónico y algunos presentaron ambas situaciones: timidina, que correspondería a la presencia del polimorfismo, y citosina, que es lo que está descrito mayoritariamente.
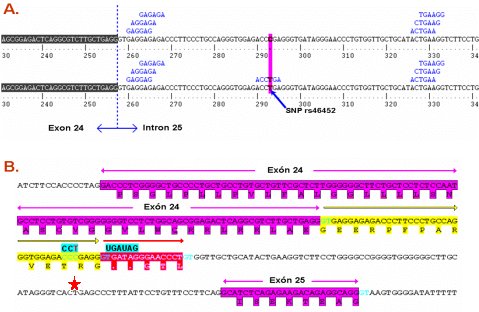
Para determinar si este cambio pudiera tener algún significado a nivel molecular, se analizaron estas secuencias en las bases de datos (Fig. 8). En la parte (A) de la figura se muestra la región correspondiente al término del exón 24 y el comienzo del intrón 25. En la situación sin polimorfismo, esta persona tiene una citosina en esa posición; En la parte (B) de la figura se muestra el caso de otro paciente en la misma secuencia de alguien que presenta el polimorfismo SNP rs# 466452 y tendría una timidina en vez de una citosina. Al existir una timidina en esa posición aparece, según distintos software, una señal que indica la presencia de un “exon splicing enhancer” o ESE, o sea, una secuencia que favorece el procesamiento de exones. Al aparecer estas secuencias podría modificarse el procesamiento de los transcritos en el gen de nefrina; el procesamiento normal se muestra en color morado: el exón 24, después vendría el intrón y luego el exón 25. Con el polimorfismo se presentan dos situaciones posibles: la primera es que se intercale la secuencia que está en amarillo, con lo que se sintetizarían 14 aminoácidos adicionales y resultaría una proteína más larga; la alternativa es que se agregue el trozo de cinco aminoácidos que se muestra en rojo y en ese caso se producirá un término de la lectura en este punto, situación en que luego de cinco aminoácidos adicionales se encuentran dos codones de término. Se terminaría la secuencia en ese punto y se obtendría una proteína truncada, con ausencia de los exones 25 a 29.
Figura 8. Alternativas de procesamiento del gen de nefrina por presencia de ESE en SNP rs#466452 (explicación en el texto)
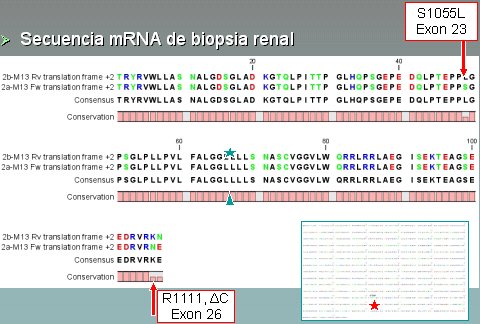
Muchas veces la bioinformática hace predicciones que indican la presencia de ciertas secuencias o elementos, sin embargo eso no significa necesariamente que dichos elementos son funcionales. Para determinar si lo observado ocurriría en la realidad, es decir para evaluar si dicho elemento es funcional se obtuvo la biopsia renal de un paciente diabético tipo 2, de 70 años de edad, con daño renal, macroalbuminúrico y portador de carcinoma renal. De esta biopsia se extrajo RNA, es decir, los transcritos, para determinar lo que ocurría en esta región del gen de nefrina en este paciente, que era portador del polimorfismo. Se esperaba encontrar un cambio en la los transcritos, que serían un reflejo de cambios en el procesamiento. Al analizar la secuencia de ambas hebras del gen de nefrina del paciente, no se encontró cambio alguno en la región del polimorfismo SNP rs# 466452, cambios que se reflejarían en la zona más terminal de la proteína. Sin embargo, al secuenciar el ADN extraído de la biopsia renal se observó que este paciente presentaba dos mutaciones en esa zona. Estas eran mutaciones distintas, nuevas, sin relación con el cambio de procesamiento ni con el polimorfismo que se estaba estudiando: en una hebra presentaba una mutación puntual, correspondiente a un cambio de un aminoácido por otro diferente en el exón 23, (una serina por una leucina); y en la otra hebra presentaba un deleción puntual de una citosina en el exón 26. Al perderse una base se produciría un corrimiento en el marco de lectura y un cambio de todos los aminoácidos siguientes. Esta persona, debiera tener una proteína con una secuencia distinta a partir de este punto que, por lo demás, termina poco después dado que aparece un codón de término de la traducción, de modo que el 50% de su nefrina sería truncada y el otro 50% tendría un cambio puntual. No se sabe si eso tiene que ver con la aparición del tumor que dio origen a intervención quirúrgica que nos permitió obtener la biopsia (Fig. 9).
Figura 9. Paciente con SNP rs#466452, de 70 años, DM 2 mal controlada, macroalbuminúrico, carcinoma renal, 2 polos de 7 cm riñón derecho
En este punto se encuentra nuestra investigación. El resto del trabajo tiene un enfoque distinto; se trata de contestar otra pregunta: por qué las personas que presentan nefropatía diabética suelen ser mucho más susceptibles a eventos cardiovasculares. Se sabe que las personas que presentan daño renal, desde la etapa de microalbuminuria, tienen mayor incidencia de infartos. De hecho, la microalbuminuria es un marcador de problemas coronarios y nos interesó buscar la relación entre ambos hechos. Esto nos llevó a analizar las zonas reguladoras de la expresión del gen de nefrina, es decir, el promotor de ese gen, y descubrir que contiene elementos que están descritos como elementos de respuesta a insulina. Lo dicho podría ser interesante desde el punto de vista de la diabetes mellitus, porque si la insulina no está en niveles normales en un paciente diabético, bien podría ser que esté afectada la expresión del gen de nefrina.
Para determinar la presencia de cambios a nivel de las zonas reguladoras del gen de nefrina en nuestros pacientes, se estudió a 62 individuos controles, entre los que se contaban 22 individuos sanos, 20 nefrópatas no diabéticos y 20 pacientes coronarios no diabéticos; Los casos corresponden a 20 pacientes diabéticos tipo 2 normoalbuminúricos sin problemas coronarios y 20 diabéticos tipo 2 macroalbuminúricos, con historia de infarto o algún episodio cardiovascular. Nuestro propósito es analizar la zona del gen correspondiente al promotor, es decir las zonas reguladoras, vecinas a las secuencias que codifican para la proteína como tal. Esta zona resultó ser más compleja de lo que se esperaba; por ejemplo, se ha descrito que existe más de un punto de inicio de la transcripción en el gen de nefrina. Es bastante común en genes eucarióticos, que existan dos promotores diferentes; se sabe que se utiliza un punto de inicio para la expresión de la nefrina en el cerebelo y otro punto de inicio para la expresión de la nefrina en riñón. Nos propusimos buscar, mediante el sistema que se utilizó para las otras regiones del gen, la presencia de polimorfismos en el promotor del gen de nefrina, en aquellas regiones que contienen señales de respuesta a insulina, señales que son secuencias GAGAGAGA. El estudio todavía está en proceso y aún no hay resultados definitivos. Lo que se ha obtenido hasta este momento se resume en la (Fig. 10) que muestra una porción pequeña de la secuencia analizada. En las zonas ricas en GAGAGAGA, hasta el momento no se encuentra nada que distinga los distintos grupos de pacientes. Las secuencias de consenso se ven todas similares.
Figura 10. Secuencia parcial del promotor del gen de nefrina
Los polimorfismos SNPs rs3814995 y rs4806213 no sinónimos del gen de nefrina están presentes en algunos pacientes diabéticos y nefrópatas no diabéticos estudiados, situación que se podría extrapolar a la población chilena, pero que no tendrían asociación con diabetes ni con nefropatía diabética. El SNP rs#466452 o polimorfismo intrónico, presente en el intrón 24, es más frecuente en diabéticos tipo 1. No hay relación entre la presencia del polimorfismo intrónico SNP rs#466452 y el grado de albuminuria en pacientes diabéticos tipo 1. La presencia de este cambio puntual SNP rs#466452 presente en el intrón no genera un cambio en el procesamiento del gen de nefrina; o sea, a pesar de que está la secuencia de consenso, no se puede decir que la secuencia sea funcional como ESE.
En este momento estamos abocados a confirmar los hallazgos descritos. También hemos desarrollado un test rápido de diagnóstico para ver la presencia del polimorfismo SNP rs#466452, que se podría utilizar en clínica, en caso de que determinemos que está asociado con alguna patología. Además tratamos de ver si detectamos nefrina en orina de pacientes. Primero ensayamos unos anticuerpos policlonales comerciales que no funcionaron y finalmente decidimos generar un nuevo anticuerpo policlonal antinefrina, con la ayuda de la empresa Biosonda, que está en etapa de evaluación. Nuestra intención es completar el estudio y seguir buscando los marcadores genéticos que podrían detectar en forma precoz la nefropatía diabética o el daño endotelial.













 Esta obra de Medwave está bajo una licencia Creative Commons Atribución-NoComercial 3.0 Unported. Esta licencia permite el uso, distribución y reproducción del artículo en cualquier medio, siempre y cuando se otorgue el crédito correspondiente al autor del artículo y al medio en que se publica, en este caso, Medwave.
Esta obra de Medwave está bajo una licencia Creative Commons Atribución-NoComercial 3.0 Unported. Esta licencia permite el uso, distribución y reproducción del artículo en cualquier medio, siempre y cuando se otorgue el crédito correspondiente al autor del artículo y al medio en que se publica, en este caso, Medwave.

La publicación de estas Actas Científicas ha sido posible gracias a una colaboración editorial entre Medwave y la Sociedad Chilena de Nefrología.
 Expositores:
Daniela Seelenfreund H.[1], Sergio Lobos C.[1], Pilar Durruty A.[2], Inés Barquín de la C.[3], Miriam Alvo A.[3]
Expositores:
Daniela Seelenfreund H.[1], Sergio Lobos C.[1], Pilar Durruty A.[2], Inés Barquín de la C.[3], Miriam Alvo A.[3]

Citación: Seelenfreund D, Lobos S, Durruty P, Barquín de la C. I, Alvo M. Nephrin and diabetic nephropathy: study of genetic polymorphisms. Medwave 2006 Sep;6(8):e2101 doi: 10.5867/medwave.2006.08.2301
Fecha de publicación: 1/9/2006

Nos complace que usted tenga interés en comentar uno de nuestros artículos. Su comentario será publicado inmediatamente. No obstante, Medwave se reserva el derecho a eliminarlo posteriormente si la dirección editorial considera que su comentario es: ofensivo en algún sentido, irrelevante, trivial, contiene errores de lenguaje, contiene arengas políticas, obedece a fines comerciales, contiene datos de alguna persona en particular, o sugiere cambios en el manejo de pacientes que no hayan sido publicados previamente en alguna revista con revisión por pares.
Aún no hay comentarios en este artículo.
Para comentar debe iniciar sesión