Key Words: psoriasis, genetics, IFIH1, polymorphism, Colombia
Resumen
Introducción
La psoriasis es una dermatosis inflamatoria crónica, con presentación clínica variable y cuya etiología involucra un componente genético importante. Alrededor del mundo se han descrito múltiples variaciones genéticas asociadas a la enfermedad. Sin embargo, en población colombiana se desconocen estas variantes. En este estudio se evalúa el polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 y su asociación con el desarrollo de psoriasis en población colombiana. Además, se caracteriza a los individuos demográfica y clínicamente.
Métodos
Se realizó un estudio observacional de casos y controles, no pareado, que incluyó 51 individuos con psoriasis y 151 controles poblacionales, todos de ancestría paisa (proveniente de la región de Antioquia) auto reportada. A todos los individuos se les realizó genotipificación del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 y se les determinó la asociación con la enfermedad. Se caracterizaron demográficamente ambos grupos y los casos clínicamente.
Resultados
Se encontró que los casos presentaron una menor frecuencia del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 en relación a los controles, 5 versus 22,67% respectivamente, con un análisis de asociación alélico. No hubo diferencias significativas en edad ni en sexo. La psoriasis vulgar fue la variante de presentación más común (78%). Alrededor de la mitad de los casos presentaron psoriasis ungular (56%), en menor frecuencia artritis psoriásica (19,6%) y el 45% de los casos tuvo alguna comorbilidad.
Conclusiones
Los resultados obtenidos confirman que los portadores del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1, presentan un riesgo disminuido de desarrollar psoriasis.
Introducción
La psoriasis es una dermatosis inflamatoria crónica cuya incidencia y prevalencia mundial varía entre 60,4 a 140 casos por cada 100 000 habitantes y de 0,09 a 5,6%, respectivamente [1].
Clínicamente se reconocen diferentes subtipos entre los que se encuentran la psoriasis vulgar, la psoriasis en gotas, la psoriasis eritrodérmica, la psoriasis palmo-plantar, la psoriasis inversa y la psoriasis pustular, que pueden o no cursar con afectación ungular y/o articular [2],[3]. La forma más común de estos fenotipos clínicos es la psoriasis vulgar, que en algunas series representa hasta el 90% de todos los casos [3]. Aunque la etiopatogenia de la psoriasis aún no se conoce completamente, se sabe que la enfermedad se manifiesta como resultado de anomalías bioquímicas, inmunológicas y del sistema vascular en individuos con cierta predisposición genética [2]. Adicionalmente, la psoriasis se ha relacionado con varias comorbilidades tales como las enfermedades inflamatorias del intestino, el síndrome cardiovascular, el síndrome metabólico, la depresión y el cáncer [4],[5].
Al igual que en cualquier otra enfermedad autoinmune, la patogenia de la psoriasis requiere la combinación de tres componentes: la predisposición genética, los factores ambientales y la alteración de la regulación inmunitaria.
La contribución de determinantes genéticos a la psoriasis, se ha podido dilucidar a partir de estudios epidemiológicos, estudios de ligamiento genético [6],[7], y más recientemente estudios de asociación del genoma completo (GWAS, Genome- Wide-Association- Studies) [6]. Estos últimos han permitido identificar polimorfismos de nucleótido único (SNP, Simple Nucleotide Polymorphism) asociados al desarrollo de la enfermedad [6],[8]. La mayoría de ellos se localizan en genes que intervienen en procesos de presentación antigénica (HLA- C y ERAP1), respuesta inmune innata antiviral (IFIH1, DDX58, TYK2, RNF114) y, esencialmente, que participan en vías de activación de la respuesta inmune Th17 (IL12B, IL23A, IL23R, TRAF3IP2 y NFKBIZ) [9],[10].
Entre los genes descritos se encuentra el IFIH1 (Interferon Induced with Helicase c domain 1) que está ubicado en el cromosoma 2q24.3. Este gen codifica para la proteína MDA5 que comprende tres dominios:
- Un dominio N- terminal de reclutamiento de caspasas (CARD, Caspase Recruitment Domain), con el cual inicia cascadas de señalización que conducen a la activación final del factor regulatorio de interferón (IRF, interferon regulatory factor) 3, IRF-7 y NF-k B.
- Un dominio central de ARN helicasas, con actividad antiviral, que desencadena la activación de una proteína adaptadora, el estimulador 1 del promotor del IFN-B (IPS-1 , IFN-B promoter stimulator- 1) y la producción de IFN-B , entre otras citoquinas proinflamatorias.
- Un dominio C- terminal [11],[12]. Los polimorfismos de nucleótido único en este gen al parecer alteran la función y/o disminuyen la concentración de la proteína MDA5 en células mononucleares circulantes, reduciendo la producción de factores pro inflamatorios involucrados en la patogénesis de enfermedades autoinmunes [13], y concediendo a su portador disminución en el riesgo de desarrollarlas [14],[15].
En un estudio realizado en nuestra población, se evaluaron variantes en el gen IFIH1, incluyendo el polimorfismo de nucleótido único rs10930046, en una cohorte de tríos familiares con diabetes mellitus tipo 1 (T1D). El alelo C de este polimorfismo de nucleótido único reveló un efecto protector para desarrollar T1D1 (205 casos versus 96 sanos) [16]. De manera interesante, en otro estudio realizado en población caucásica, este alelo también mostró el mismo efecto en la psoriasis (2098 casos versus 1744 controles) [10].
Hasta la fecha en Colombia no se han encontrado reportes de estudios genéticos relacionados con la psoriasis. Es así como el antecedente encontrado del polimorfismo de nucleótido único rs10930046 (gen IFIH1) como alelo protector para la diabetes tipo 1 en población colombiana [16] nos alentó a dilucidar el efecto de este en la psoriasis, al estar implicada la autoinmunidad en la fisiopatogenia de ambas enfermedades. Por tal motivo, el objetivo de este estudio fue el evaluar la asociación del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 con el desarrollo de la psoriasis en pacientes colombianos.
Métodos
Población
El diseño del estudio fue de tipo de casos y controles no pareados. La relación de casos y controles fue de 1:3. Los casos correspondieron a pacientes con psoriasis recolectados de forma prospectiva (casos incidentes), durante la atención dermatológica en diferentes centros de Medellín-C olombia. Los criterios de inclusión de los casos fueron tener:
- Cualquier variante clínica de psoriasis, diagnosticada clínica y/o histopatológicamente por un dermatólogo.
- Ancestría paisa (población colombiana bien caracterizada y estudiada genéticamente, predominante en la región de Antioquia) auto reportada.
- Edad igual o mayor a 18 años.
Los criterios de exclusión de los casos correspondieron a cualquier déficit mental que impidiera su participación en el estudio.
Todos los pacientes firmaron un consentimiento informado. El estudio se enmarcó en las normas de investigación en seres humanos estipuladas en la resolución N° 8430 de 1993 del Ministerio de Salud de Colombia y en la Declaración de Helsinki de 2013. El estudio fue sometido y aprobado por el comité de ética de la Institución Prestadora de Servicios de Salud Universitaria, el Hospital San Vicente Fundación y del Instituto de Investigaciones Médicas de la Facultad de Medicina de la Universidad de Antioquia, en Medellín, Colombia. En los casos se verificó la ancestría paisa hasta el segundo grado de consanguinidad, y se recolectó información sociodemográfica y clínica. Cada paciente fue evaluado por dermatólogo. Se definió como psoriasis de inicio temprano, aquella que se presentaba antes de los 40 años. Para la evaluación de la gravedad de la psoriasis se usó la escala clínica índice de la severidad del área de psoriasis (Psoriasis Area and Severity Index, PASI). La psoriasis ungular fue definida por las alteraciones inherentes de la enfermedad en la placa ungular, en una o más uñas. También se evaluó la presencia de artritis psoriásica. Para el antecedente familiar de psoriasis se consideraron tres grupos: primer, segundo y tercer o mayor grado de consanguinidad.
Los controles correspondieron a individuos de ancestría paisa auto reportada, padres de niños con diagnóstico de epilepsia idiopática generalizada, sin antecedentes de enfermedades autoinmunes, y que habían participado en un estudio previo de genética local. La información demográfica y genética (específicamente la presencia o ausencia del polimorfismo de nucleótido único rs10930046 – p. His460Arg – en el gen IFIH1) se obtuvo de dicho registro. Entre los controles se excluyeron quienes tenían el antecedente de cualquier enfermedad autoinmune, y pacientes en quienes se identificará el diagnóstico de psoriasis en el registro genético.
selección del Polimorfismo de nucleótido único
Se seleccionó el polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1, localizado en el cromosoma 2q24.3, en el cual se presenta una sustitución de cambio de sentido (missense) en la proteína llevando al remplazo de histidina por arginina en la posición 460 (disponible en http:/www. genome.u csc.edu). Su elección se basó en el estudio publicado en Norteamérica relacionándolo con la psoriasis como factor protector en norteamericanos caucásicos [10], pero también porque este mismo polimorfismo se ha asociado al desarrollo de otras enfermedades inflamatorias y autoinmunes [12],[16].
Genotipificación
El ADN fue extraído a partir de células bucales usando el método de salting out modificado [17]. La genotipificación se realizó por el método tetraprimer ARMS-P CR (Amplification Refractory Mutation System- Polymerase Chain Reaction), en un termociclador Eppendorf Nexus X2. Los cebadores utilizados fueron: 5’- TTTT AGCT TTGC TTTCA TTGGTTATACA- 3’ (delantero externo), 5’-T GTTTTTCA ACT TCTG CATC AAATCAT3’ (delantero interno), 5’-A AAG CTAT TTTCT CAC TGTA GTGTGCTG-3 ’ (trasero externo) y 5’-GAAG CAGT GTAT AATA ACAT CATG AGTCG-3 ’ (trasero interno). Las condiciones de la PCR fueron: 94 grados Celsius por cinco minutos; 35 ciclos de 94 grados Celsius por 45 segundos; el alineamiento se realizó a 56 grados Celsius por 45 segundos seguido de una extensión a 72 grados Celsius por 45 segundos; y el paso de extensión final a 72 grados Celsius durante 10 minutos. Losproductos de la PCR fueron directamente analizados por electroforesisen gel de agarosa al 3%, teñido con EZVision(AMRESCO, SOLON, OH, USA) y cada alelo fue reconocidode acuerdo con el tamaño (Figura 1).
Tamaño de muestra
De acuerdo con lo reportado por Rodríguez y colaboradores [16], y basados en una prevalencia de 0,31 del polimorfismos denucleótido único 460Arg (rs10930046) en el gen IFIH1, se realizóun cálculo de muestra considerando un poder del 80%, unnivel de confianza del 90%, un Odds ratio esperable de 0,41 [16],lo que arrojó una muestra mínima de 45 casos y 135 controles(OpenEpi© Version 3.03.17) [18].
Análisis estadístico
Se realizó un análisis univariado de las variables para conocersus frecuencias. La distribución normal de las variables cuantitativas se evaluó con la prueba de Kolmogorof-Smirnov. Mediante análisis de regresión logística multivariada se identificaron asociaciones significativas y la fuerza de asociación entre las variables de exposición y respuesta, además de la verificaciónde potenciales confusores (por ejemplo, presencia de autoinmunidad). En tal sentido, se estimó la medida de asociación usual en los estudios de casos y controles (el Odds ratio o razón de disparidad), acompañado de su respectivo intervalo de confianza del 95%. El software PLINK versión 1.9, se usó para calcular las frecuencias alélicas, el equilibrio de Hardy-Weinberg(HWE, Hardy-Weinberg Equilibrium) y la asociación alélica delrs10930046 con la psoriasis. Fue considerado estadísticamente significativo un valor de p < 0,05.
Caracterización demográfica
Todos los individuos del estudio tuvieron ancestría paisa autoreportada. La población “paisa” corresponde a una distribución étnica especial en Colombia que presenta una alta prevalenciade consanguinidad y aislamiento geográfico. La edad promedio de los casos y controles fue de 53 años (desviación estándar ± 16,4) y de 47,5 años (desviación estándar ±14,7), respectivamente. En el grupo de los casos los hombres representaron el 45% de la muestra, sin encontrar diferencias significativas entre hombres y mujeres. No se encontraron diferencias estadísticamente significativas en el sexo o en la edad (Tabla 1).
Caracterización clínica de los casos
Entre los casos, la edad al inicio y diagnóstico de la enfermedadfue de 37,8 (desviación estándar ± 19) y 40 (desviación estándar ± 18,9) años, respectivamente. La psoriasis de inicio temprano correspondió al 60,7% de los casos (48% hombres y 51% mujeres). La variante clínica más común fue la psoriasis vulgar (Tabla 2). En cuanto a la clasificación de la gravedad de la psoriasis, la forma leve a moderada fue la más frecuente (80% delos casos de psoriasis: 41/51).
La psoriasis ungular se encontró en 56% (27/51) de los casos yfue más común la presencia de cambios ungulares sin artritis psoriásica que con ella. Sin embargo, de los 19,6% (10/51) pacientes que reportaron diagnóstico de artritis psoriásica, la mayoría (60%; 6/10) presentó concomitantemente alteraciones ungulares atribuibles a psoriasis.
Tal y como se describe en la Tabla 3, las comorbilidades más frecuentemente encontradas en la población estudiada correspondierona la hipertensión arterial (HTA) y la diabetes mellitustipo 2. Se encontró historia familiar de psoriasis en el 45% (23/51) de los casos, y de estos el 43, 4 y 47% fueron en primer, segundo y tercer o mayor grado de consanguinidad, respectivamente. En lo referente a la autoinmunidad se encontró que 29% (15/51) de los casos presentaron antecedentes familiares de lupuseritematoso sistémico, artritis reumatoide, dermatomiositis y/o hipotiroidismo autoinmune.
Tabla 1. Caracterización de los casos y los controles.
Tabla 2. Variantes clínicas de psoriasis en los casos.
Tabla 3. Comorbilidades en pacientes con psoriasis.
Análisis genético
La tasa de genotipificación fue del 99% tanto para los casos(50/51 muestras) como para los controles (150/151 muestras). En la Tabla 4 se exponen las frecuencias genotípicas encontradaspara el marcador rs10930046 en el gen IFIH1.
Tal y como se observa en la Tabla 4, el marcador estuvo enequilibrio de Hardy-Weinbergen el grupo control. El análisis deasociación alélica del alelo C del rs 10930046 con psoriasis fueestadísticamente significativo (Odds ratio: 0,17; intervalo de confianza95%: 0,07 a 0,45) y un valor de p < 0,05 con lo que sedemuestra que este alelo confiere a su portador, protección significativafrente al desarrollo de la psoriasis.
Para ratificar los resultados, se realizó un segundo análisis deasociación alélica, comparando los casos con los genotipos de96 individuos sanos, mayores de 18 años, de ancestría paisa,disponibles públicamente en la base de datos 1000 genomas [19]. Aun en este análisis se observó asociación significativa delalelo C con psoriasis (Odds ratio: 0,17, intervalo de confianza95%:0,06 a 0,46) y un valor de p < 0,05 (Tabla 5).
Tabla 4. Genotipificación del marcador rs10930046 (His460Arg) en el gen IFIH1.
Tabla 5. Asociación alélica entre los casos y los controles.
Discusión
Los resultados presentados sugieren que el polimorfismos de nucleótido único rs10930046 en el gen IFIH1 modifica el riesgo de desarrollar psoriasis en la población colombiana con ancestría paisa, encontrándose una similitud de las características clínicas y demográficas de nuestra población con las reportadas en los pacientes con psoriasis en Colombia [20] y alrededor del mundo [21],[22].
Al igual que lo encontrado en este estudio, la psoriasis afecta predominantemente a la población mayor de 18 años, siendo más común desde los 30 hasta los 39 años y de los 50 a los 69 años [21],[23], con un inicio temprano en el 65,2% de los casos [24]. La psoriasis vulgar fue la variante más común. Además, se encontraron similitudes de las características clínicas y demográficas de nuestra población con las reportadas en los pacientes con psoriasis en Colombia [20] y alrededor del mundo [1],[3],[21],[22].
En Colombia, y en general en Latinoamérica, no se han encontrado reportes de estudios poblacionales de prevalencia de la psoriasis. Un estudio previo encontró una prevalencia global de artritis psoriásica de 19,7% y en Latinoamérica de 21,5% [1]. Estos datos son similares a los encontrados en nuestro estudio en donde 19,6% tenían esta condición. Con respecto a la psoriasis ungular, se ha reportado en cerca del 50% de los pacientes con psoriasis [25], lo que se asemeja al 56% encontrado en este estudio.
En Colombia, estudios realizados en Bogotá reportaron edades medias de diagnóstico de 46,3 y 54,5 años, con distribución similar entre hombres y mujeres [20]. Estos datos de epidemiología local, son comparables con los reportados en el ámbito mundial, excepto para la artritis psoriásica donde se expone una frecuencia baja. Lo anterior pudiera explicarse por el posible subdiagnóstico, dato que se puede contrastar con los hallazgos del presente trabajo, en el que los pacientes tuvieron prevalencias de artritis psoriásica similares a las reportadas en otras partes del mundo y en estudios latinoamericanos [26].
Desde el punto de vista genético, son múltiples los estudios que han llevado a identificar variantes genéticas en los afectados, que en su mayoría codifican para moléculas que participan enprocesos inmunológicos estrechamente relacionados con la enfermedad [27] En el gen IFIH1 (MDA5) se han descrito los siguientes polimorfismos de nucleótido único relacionados a lapsoriasis: rs17716942 [28], rs1990760 y rs35667974 [29],rs17715343 y rs13431841 [30].
Específicamente con el gen IFIH1 solo se ha reportado un estudio previo que reporta la asociación entre el polimorfismos de nucleótido único rs10930046 y el desarrollo de la psoriasis [10]. Dicho hallazgo se asemeja a lo encontrado en el presente estudio, confirmándose así el papel protector para psoriasis que este polimorfismo de nucleótido único confiere a sus portadores en estas dos poblaciones específicas, las que pudieran tener algunas semejanzas genéticas que explicarían estos resultados. Adicionalmente, el hecho de haber encontrado un rol protector del mismo polimorfismo de nucleótido único para la diabetes mellitus tipo 1 en esta misma población apoyaría la hipótesis de una etiología compartida para diferentes formas de presentaciónde procesos de autoinmunidad e inflamación [16].
Una de las limitaciones encontradas en el estudio fue la falta de información con respecto a la presencia o ausencia de psoriasis en los controles. Además, estos correspondieron a padres de niños con epilepsia idiopática. Otra limitación fue un tamaño muestral insuficiente para encontrar posibles asociaciones con las variantes clínicas de la enfermedad y las comorbilidades. No obstante, el poder de la muestra fue suficiente para establecer la asociación genética protectora del polimorfismo de nucleótido único rs10930046 en el gen IFIH1 en los pacientes con psoriasis en nuestra población, la cual correspondió a nuestra hipótesis de trabajo.
Entre las fortalezas de estudio se destacan la rigurosidad con laque se realizaron tanto las pruebas genéticas como la alta calidad metodológica del estudio, que permitió disminuir al máximo los sesgos relacionados con los estudios de casos y controles.
Conclusión
Se presenta el primer estudio de genética en psoriasis realizado en población paisa colombiana, en el que, mediante un análisisde asociación alélico de casos y controles, se confirmó que los portadores del polimorfismo de nucleótido único rs10930046(p. His460Arg) en el gen IFIH1 tienen un menor riesgo a desarrollar la psoriasis.
Estos resultados sugieren que a futuro se haría necesarioaumentar el tamaño muestral. De esta manera se podrían contrastarlos hallazgos genéticos con las variables demográficas y clínicas, teniendo en cuenta la baja frecuencia del alelo C en la población de estudio, tal y como se ha podido demostrar en otros estudios genéticos que se han enfocado en el complejo mayor de histocompatibilidad [9],[31].
Por último, estudios de asociación genética como este permiten determinar características relacionadas con el endotipo de la psoriasis. Ello facilitara la identificación y desarrollo futuro de terapias eficaces para pacientes con determinadas características genéticas. Lo anterior no solo beneficiaría a los pacientes sino también a los sistemas de salud, ya que se garantizaría que cada paciente reciba el tratamiento adecuado para su psoriasis.
Notas
Autoría
EVEL, CJES, DMCS: conceptualización, trabajo delaboratorio, evaluación de pacientes, análisis de resultados, escritura, revisión y edición de artículo.GS: conceptualización,metodología, evaluación de pacientes, análisis de resultados,software, visualización, supervisión, administración delproyecto, obtención de financiación, escritura, revisión yedición de artículo. NGP: conceptualización, metodología,trabajo de laboratorio, análisis de resultados, software,visualización, supervisión, administración del proyecto,obtención de financiación, escritura, revisión y edición de artículo.
Agradecimientos
A Blear Holguín, auxiliar del laboratorio del Grupo de Investigación Mapeo Genético de la Universidad de Antioquia, a Diana Cornejo, estudiante de Doctorado de la Corporación de Ciencias Básicas Biomédicas de la Universidad de Antioquia. Especiales agradecimientos a Mateo Cuervo, Daniela Gómez, Manuel Martínez y Natalia Vélez (residentes de Dermatologíade la Universidad de Antioquia), y al Servicio de Fototerapia y Fotodermatología de la Institución Prestadora de Servicios de Salud de la Universidad de Antioquia. Por último, agradecemos a Alejandra Venegas, auxiliar de investigación del Grupo de Investigación Dermatológica (GRID) de la Universidad de Antioquia por su colaboración en contactar a los pacientes y en la gestión del consentimiento informado.
Conflictos de intereses
Los autores completaron la declaración de conflictos deinterés de ICMJE y declararon que no recibieron fondos por la realización de este artículo; no tienen relaciones financieras con organizaciones que puedan tener interés en el artículo publicado en los últimos tres años y no tienen otras relacioneso actividades que puedan influenciar en la publicación del artículo. Los formularios se pueden solicitar contactando alautor responsable o al Comité Editorial de la Revista.
Financiamiento
El estudio fue financiado con recursos del Grupo de Mapeo Genético y del Grupo de Investigación Dermatológica (GRID), adscritos ambos al Instituto de Investigaciones Médicas de la Facultad de Medicina de la Universidad de Antioquia, Medellín, Colombia.
Aspectos éticos
Este estudio fue aprobado por el comité de ética de la InstituciónPrestadora de Servicios de Salud Universitaria, el Hospital SanVicente Fundación y del Instituto de Investigaciones Médicasde la Facultad de Medicina de la Universidad de Antioquia enMedellín, Colombia.
Declaración de acceso a datos
La base de datos de este estudio se encuentra disponible asolicitud del interesado.
Origen y arbitraje
No solicitado. Con revisión por pares externa, por tres árbitrosa doble ciego.
Idioma del envío
Español.

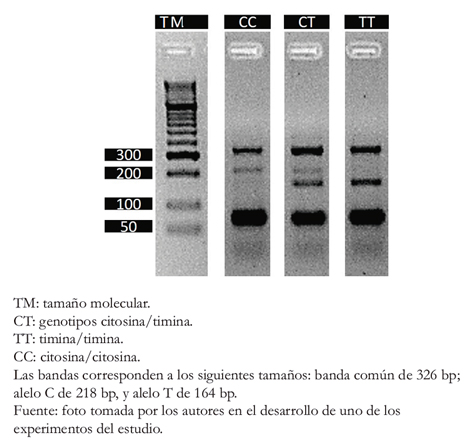 Figura 1. Electroforesis en gel de agarosa del producto de amplificación del rs10930046 (p. His460Arg) en el gen IFIH1
Figura 1. Electroforesis en gel de agarosa del producto de amplificación del rs10930046 (p. His460Arg) en el gen IFIH1

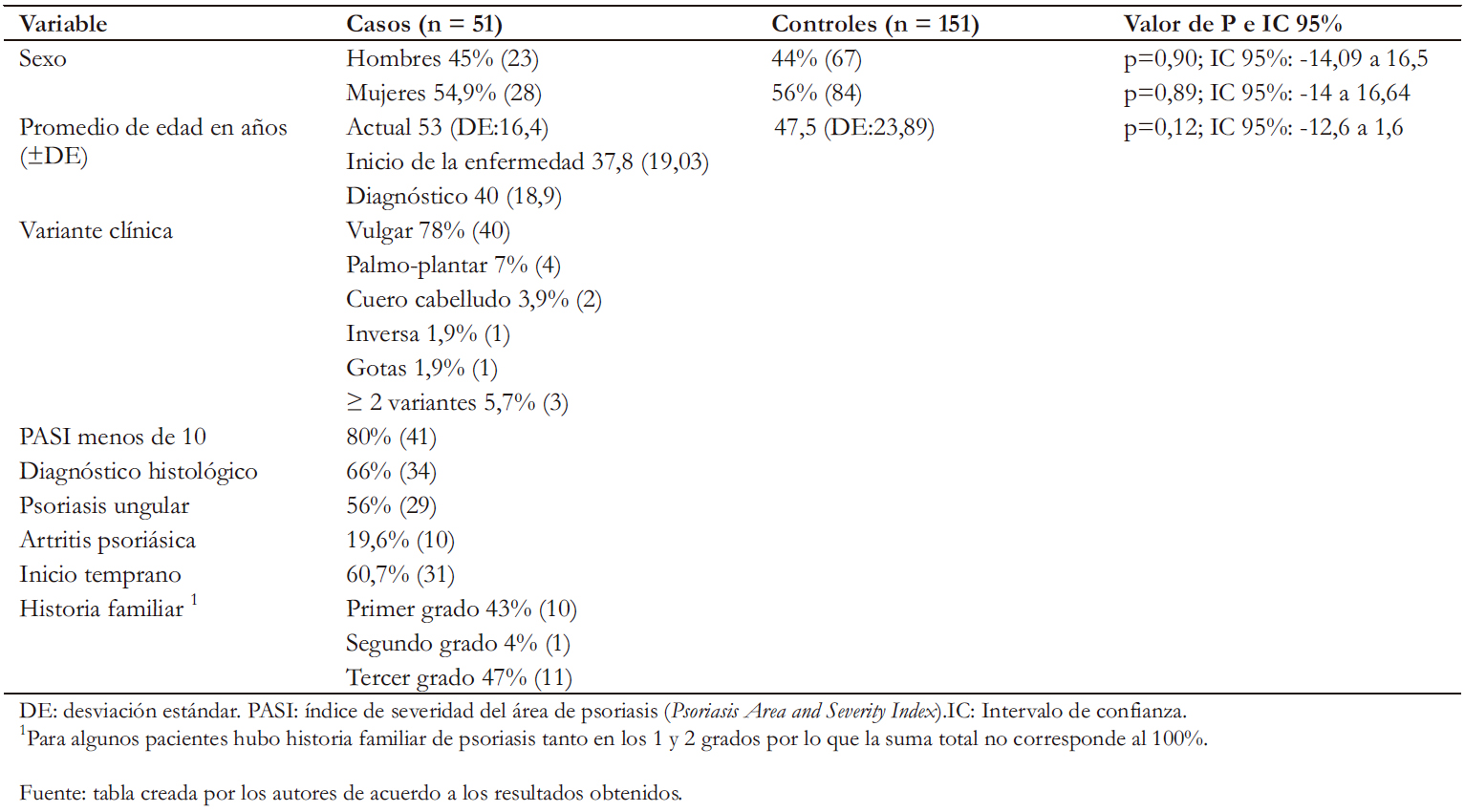 Tabla 1. Caracterización de los casos y los controles.
Tabla 1. Caracterización de los casos y los controles.

 Tabla 2. Variantes clínicas de psoriasis en los casos.
Tabla 2. Variantes clínicas de psoriasis en los casos.

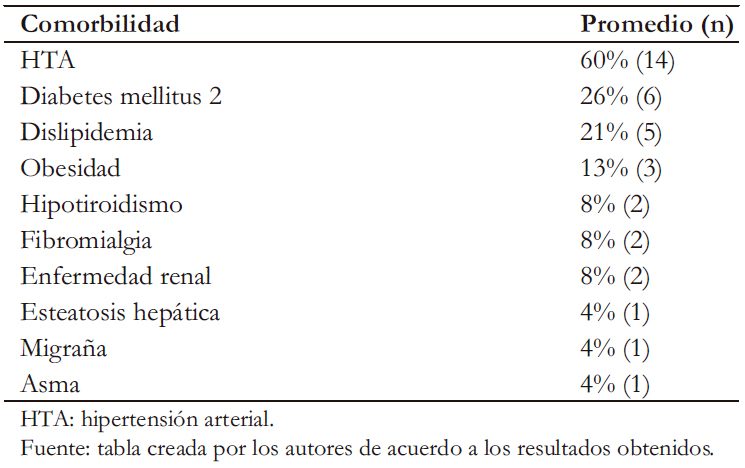 Tabla 3. Comorbilidades en pacientes con psoriasis.
Tabla 3. Comorbilidades en pacientes con psoriasis.

 Tabla 4. Genotipificación del marcador rs10930046 (His460Arg) en el gen IFIH1.
Tabla 4. Genotipificación del marcador rs10930046 (His460Arg) en el gen IFIH1.

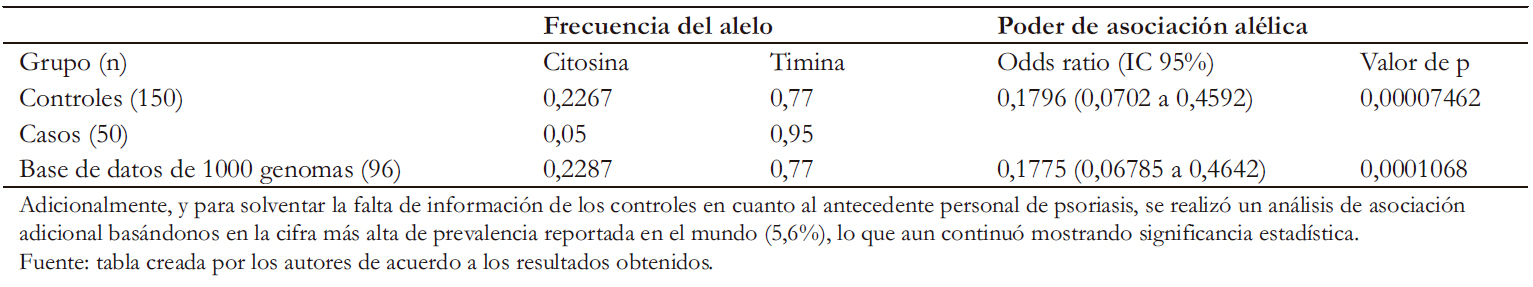 Tabla 5. Asociación alélica entre los casos y los controles.
Tabla 5. Asociación alélica entre los casos y los controles.

Introducción
La psoriasis es una dermatosis inflamatoria crónica, con presentación clínica variable y cuya etiología involucra un componente genético importante. Alrededor del mundo se han descrito múltiples variaciones genéticas asociadas a la enfermedad. Sin embargo, en población colombiana se desconocen estas variantes. En este estudio se evalúa el polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 y su asociación con el desarrollo de psoriasis en población colombiana. Además, se caracteriza a los individuos demográfica y clínicamente.
Métodos
Se realizó un estudio observacional de casos y controles, no pareado, que incluyó 51 individuos con psoriasis y 151 controles poblacionales, todos de ancestría paisa (proveniente de la región de Antioquia) auto reportada. A todos los individuos se les realizó genotipificación del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 y se les determinó la asociación con la enfermedad. Se caracterizaron demográficamente ambos grupos y los casos clínicamente.
Resultados
Se encontró que los casos presentaron una menor frecuencia del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1 en relación a los controles, 5 versus 22,67% respectivamente, con un análisis de asociación alélico. No hubo diferencias significativas en edad ni en sexo. La psoriasis vulgar fue la variante de presentación más común (78%). Alrededor de la mitad de los casos presentaron psoriasis ungular (56%), en menor frecuencia artritis psoriásica (19,6%) y el 45% de los casos tuvo alguna comorbilidad.
Conclusiones
Los resultados obtenidos confirman que los portadores del polimorfismo de nucleótido único rs10930046 (His460Arg) en el gen IFIH1, presentan un riesgo disminuido de desarrollar psoriasis.
 Authors:
Evelyn Vanesa Erazo Luna[1,2], Claudia Janeth Echavarría Sierra[1,2], Diana M. Cornejo-Sánchez[2], Gloria Sanclemente[1], Nicolás Guillermo Pineda Trujillo[2]
Authors:
Evelyn Vanesa Erazo Luna[1,2], Claudia Janeth Echavarría Sierra[1,2], Diana M. Cornejo-Sánchez[2], Gloria Sanclemente[1], Nicolás Guillermo Pineda Trujillo[2]
Affiliation:
[1] Grupo de Investigación Dermatológica (GRID), Instituto de Investigaciones Médicas, Universidad de Antioquia, Medellín, Colombia
[2] Grupo de Mapeo Genético, Instituto de Investigaciones Médicas, Universidad de Antioquia, Medellín, Colombia
E-mail: gloria.sanclemente@udea.edu.co
Author address:
[1] Carrera 25 A # 1 A Sur 45, Of 2026
Torre Médica El Tesoro
Medellín, Colombia

Citation: Erazo Luna EV, Echavarría Sierra CJ, Cornejo-Sánchez DM, Sanclemente G, Pineda Trujillo NG. Protective association exhibited by a single nucleotide polymorphism of the IFIH1 gene in patients with psoriasis: A case-control study. Medwave 2021;21(11):e002099 doi: 10.5867/medwave.2021.11.002099
Submission date: 13/2/2021
Acceptance date: 31/8/2021
Publication date: 3/12/2021
Origin: Not commissioned
Type of review: Externally peer-reviewed by three reviewers, double-blind

Comments (0)
We are pleased to have your comment on one of our articles. Your comment will be published as soon as it is posted. However, Medwave reserves the right to remove it later if the editors consider your comment to be: offensive in some sense, irrelevant, trivial, contains grammatical mistakes, contains political harangues, appears to be advertising, contains data from a particular person or suggests the need for changes in practice in terms of diagnostic, preventive or therapeutic interventions, if that evidence has not previously been published in a peer-reviewed journal.
No comments on this article.
To comment please log in
 Medwave provides HTML and PDF download counts as well as other harvested interaction metrics.
Medwave provides HTML and PDF download counts as well as other harvested interaction metrics. There may be a 48-hour delay for most recent metrics to be posted.

- Hernández-Vásquez A, Molinari L, Larrea N, Ciapponi A. Psoriasis in Latin America and the Caribbean: a systematic review. J Eur Acad Dermatol Venereol. 2017;31(12):1991-8. | CrossRef |
- Armesto S, Esteve A, Coto-Segura P, Drake M, Galache C, Martínez-Borra J, et al. Nail psoriasis in individuals with psoriasis vulgaris: a study of 661 patients. Actas Dermosifiliogr. 2011;102(5):365-72. | CrossRef |
- Naldi L, Gambini D. The clinical spectrum of psoriasis. Clin Dermatol. 2007;25(6):510-8. | CrossRef |
- Takeshita J, Grewal S, Langan SM, Mehta NN, Ogdie A, Van Voorhees AS, et al. Psoriasis and comorbid diseases: Epidemiology. J Am Acad Dermatol. 2017;76(3):377-90. | CrossRef |
- Dowlatshahi EA, Wakkee M, Arends LR, Nijsten T. The prevalence and odds of depressive symptoms and clinical depression in psoriasis patients: a systematic review and meta-analysis. J Invest Dermatol. 2014;134(6):1542-51. | CrossRef |
- Capon F. The Genetic Basis of Psoriasis. Int J Mol Sci. 2017;18(12). | CrossRef |
- Puig L, Julià A, Marsal S. The pathogenesis and genetics of psoriasis. Actas Dermosifiliogr. 2014;105(6):535-45. | CrossRef |
- O'Rielly DD, Rahman P. Genetic, Epigenetic and Pharmacogenetic Aspects of Psoriasis and Psoriatic Arthritis. Rheum Dis Clin North Am. 2015;41(4):623-42. | CrossRef |
- Aterido A, Julià A, Ferrándiz C, Puig L, Fonseca E, Fernández-López E, et al. Genome-Wide Pathway Analysis Identifies Genetic Pathways Associated with Psoriasis. J Invest Dermatol. 2016;136(3):593-602. | CrossRef |
- Li Y, Liao W, Cargill M, Chang M, Matsunami N, Feng BJ, et al. Carriers of rare missense variants in IFIH1 are protected from psoriasis. J Invest Dermatol. 2010;130(12):2768-72. | CrossRef |
- Loo YM, Gale M. Immune signaling by RIG-I-like receptors. Immunity. 2011;34(5):680-92. | CrossRef |
- Bouças AP, Oliveira FoS, Canani LH, Crispim D. The role of interferon induced with helicase C domain 1 (IFIH1) in the development of type 1 diabetes mellitus. Arq Bras Endocrinol Metabol. 2013;57(9):667-76. | CrossRef |
- Chistiakov DA. Interferon induced with helicase C domain 1 (IFIH1) and virus-induced autoimmunity: a review. Viral Immunol. 2010;23(1):3-15. | CrossRef |
- Yang H, Wang Z, Xu K, Gu R, Chen H, Yu D, et al. IFIH1 gene polymorphisms in type 1 diabetes: genetic association analysis and genotype-phenotype correlation in Chinese Han population. Autoimmunity. 2012;45(3):226-32. | CrossRef |
- Nejentsev S, Walker N, Riches D, Egholm M, Todd JA. Rare variants of IFIH1, a gene implicated in antiviral responses, protect against type 1 diabetes. Science. 2009;324(5925):387-9. | CrossRef |
- Rodríguez A, Alfaro JM, Balthazar V, Pineda Trujillo N. Association analysis of PTPN22, CTLA4 and IFIH1 genes with type 1 diabetes in Colombian families. J Diabetes. 2015;7(3):402-10. | CrossRef |
- Sadeghi R, Jahani F. Salting-in and salting-out of water-soluble polymers in aqueous salt solutions. J Phys Chem B. 2012;116(17):5234-41. | CrossRef |
- Kelsey J, Whittemore A, Evans A, Thompson W. Methods in Observational Epidemiology. Second edition ed. Oxford, UK: Oxford University Press;1996.
- The International Genome Sample Resource. IGSR: The International Genome Sample Resource. 2018. | Link |
- Medina-Trujillo D. Estudio epidemiológico de los pacientes con diagnóstico de psoriasis en el Centro Dermatológico Federico Lleras Acosta (CDFLLA) del año 2011 a 2013. Bogotá, D.C.: Universidad Nacional de Colombia, Facultad de Medicina, Departamento de Medicina Interna, Programa de Dermatología; 2016. | Link |
- Parisi R, Symmons DP, Griffiths CE, Ashcroft DM, team IaMoPaACIp. Global epidemiology of psoriasis: a systematic review of incidence and prevalence. J Invest Dermatol. 2013;133(2):377-85. | CrossRef |
- Boehncke WH, Schön MP. Psoriasis. Lancet. 2015;386(9997):983-994. | CrossRef |
- Fernández-Armenteros JM, Gómez-Arbonés X, Buti-Solé M, Betriu-Bars A, Sanmartin-Novell V, Ortega-Bravo M, et al. Epidemiology of Psoriasis. A Population-Based Study. Actas Dermosifiliogr. 2018;110(5):385-392. | CrossRef |
- Chularojanamontri L, Kulthanan K, Suthipinittharm P, Jiamton S, Wongpraparut C, Silpa-Archa N, et al. Clinical differences between early- and late-onset psoriasis in Thai patients. Int J Dermatol. 2015;54(3):290-4. | CrossRef |
- Tan ES, Chong WS, Tey HL. Nail psoriasis: a review. Am J Clin Dermatol. 2012;13(6):375-88. | CrossRef |
- Alinaghi F, Calov M, Kristensen LE, Gladman DD, Coates LC, Jullien D, et al. Prevalence of psoriatic arthritis in patients with psoriasis: A systematic review and meta-analysis of observational and clinical studies. J Am Acad Dermatol. 2019;80(1):251-65.e19. | CrossRef |
- Ray-Jones H, Eyre S, Barton A, Warren RB. One SNP at a Time: Moving beyond GWAS in Psoriasis. J Invest Dermatol. 2016;136(3):567-73. | CrossRef |
- Zhang XJ, Huang W, Yang S, Sun LD, Zhang FY, Zhu QX, et al. Psoriasis genome-wide association study identifies susceptibility variants within LCE gene cluster at 1q21. Nat Genet. 2009;41(2):205-10. | CrossRef |
- Strange A, Capon F, Spencer CC, Knight J, Weale ME, Allen MH, et al. A genome-wide association study identifies new psoriasis susceptibility loci and an interaction between HLA-C and ERAP1. Nat Genet. 2010;42(11):985-90. | CrossRef |
- Yin X, Low HQ, Wang L, Li Y, Ellinghaus E, Han J, et al. Genome-wide meta-analysis identifies multiple novel associations and ethnic heterogeneity of psoriasis susceptibility. Nat Commun. 2015;6:6916. | CrossRef |
- van Vugt LJ, van den Reek JMPA, Coenen MJH, de Jong EMGJ. A systematic review of pharmacogenetic studies on the response to biologics in patients with psoriasis. Br J Dermatol. 2018;178(1):86-94. | CrossRef |
 Hernández-Vásquez A, Molinari L, Larrea N, Ciapponi A. Psoriasis in Latin America and the Caribbean: a systematic review. J Eur Acad Dermatol Venereol. 2017;31(12):1991-8. | CrossRef |
Hernández-Vásquez A, Molinari L, Larrea N, Ciapponi A. Psoriasis in Latin America and the Caribbean: a systematic review. J Eur Acad Dermatol Venereol. 2017;31(12):1991-8. | CrossRef | Armesto S, Esteve A, Coto-Segura P, Drake M, Galache C, Martínez-Borra J, et al. Nail psoriasis in individuals with psoriasis vulgaris: a study of 661 patients. Actas Dermosifiliogr. 2011;102(5):365-72. | CrossRef |
Armesto S, Esteve A, Coto-Segura P, Drake M, Galache C, Martínez-Borra J, et al. Nail psoriasis in individuals with psoriasis vulgaris: a study of 661 patients. Actas Dermosifiliogr. 2011;102(5):365-72. | CrossRef | Naldi L, Gambini D. The clinical spectrum of psoriasis. Clin Dermatol. 2007;25(6):510-8. | CrossRef |
Naldi L, Gambini D. The clinical spectrum of psoriasis. Clin Dermatol. 2007;25(6):510-8. | CrossRef | Takeshita J, Grewal S, Langan SM, Mehta NN, Ogdie A, Van Voorhees AS, et al. Psoriasis and comorbid diseases: Epidemiology. J Am Acad Dermatol. 2017;76(3):377-90. | CrossRef |
Takeshita J, Grewal S, Langan SM, Mehta NN, Ogdie A, Van Voorhees AS, et al. Psoriasis and comorbid diseases: Epidemiology. J Am Acad Dermatol. 2017;76(3):377-90. | CrossRef | Dowlatshahi EA, Wakkee M, Arends LR, Nijsten T. The prevalence and odds of depressive symptoms and clinical depression in psoriasis patients: a systematic review and meta-analysis. J Invest Dermatol. 2014;134(6):1542-51. | CrossRef |
Dowlatshahi EA, Wakkee M, Arends LR, Nijsten T. The prevalence and odds of depressive symptoms and clinical depression in psoriasis patients: a systematic review and meta-analysis. J Invest Dermatol. 2014;134(6):1542-51. | CrossRef | Puig L, Julià A, Marsal S. The pathogenesis and genetics of psoriasis. Actas Dermosifiliogr. 2014;105(6):535-45. | CrossRef |
Puig L, Julià A, Marsal S. The pathogenesis and genetics of psoriasis. Actas Dermosifiliogr. 2014;105(6):535-45. | CrossRef | O'Rielly DD, Rahman P. Genetic, Epigenetic and Pharmacogenetic Aspects of Psoriasis and Psoriatic Arthritis. Rheum Dis Clin North Am. 2015;41(4):623-42. | CrossRef |
O'Rielly DD, Rahman P. Genetic, Epigenetic and Pharmacogenetic Aspects of Psoriasis and Psoriatic Arthritis. Rheum Dis Clin North Am. 2015;41(4):623-42. | CrossRef | Aterido A, Julià A, Ferrándiz C, Puig L, Fonseca E, Fernández-López E, et al. Genome-Wide Pathway Analysis Identifies Genetic Pathways Associated with Psoriasis. J Invest Dermatol. 2016;136(3):593-602. | CrossRef |
Aterido A, Julià A, Ferrándiz C, Puig L, Fonseca E, Fernández-López E, et al. Genome-Wide Pathway Analysis Identifies Genetic Pathways Associated with Psoriasis. J Invest Dermatol. 2016;136(3):593-602. | CrossRef | Li Y, Liao W, Cargill M, Chang M, Matsunami N, Feng BJ, et al. Carriers of rare missense variants in IFIH1 are protected from psoriasis. J Invest Dermatol. 2010;130(12):2768-72. | CrossRef |
Li Y, Liao W, Cargill M, Chang M, Matsunami N, Feng BJ, et al. Carriers of rare missense variants in IFIH1 are protected from psoriasis. J Invest Dermatol. 2010;130(12):2768-72. | CrossRef | Bouças AP, Oliveira FoS, Canani LH, Crispim D. The role of interferon induced with helicase C domain 1 (IFIH1) in the development of type 1 diabetes mellitus. Arq Bras Endocrinol Metabol. 2013;57(9):667-76. | CrossRef |
Bouças AP, Oliveira FoS, Canani LH, Crispim D. The role of interferon induced with helicase C domain 1 (IFIH1) in the development of type 1 diabetes mellitus. Arq Bras Endocrinol Metabol. 2013;57(9):667-76. | CrossRef | Chistiakov DA. Interferon induced with helicase C domain 1 (IFIH1) and virus-induced autoimmunity: a review. Viral Immunol. 2010;23(1):3-15. | CrossRef |
Chistiakov DA. Interferon induced with helicase C domain 1 (IFIH1) and virus-induced autoimmunity: a review. Viral Immunol. 2010;23(1):3-15. | CrossRef | Yang H, Wang Z, Xu K, Gu R, Chen H, Yu D, et al. IFIH1 gene polymorphisms in type 1 diabetes: genetic association analysis and genotype-phenotype correlation in Chinese Han population. Autoimmunity. 2012;45(3):226-32. | CrossRef |
Yang H, Wang Z, Xu K, Gu R, Chen H, Yu D, et al. IFIH1 gene polymorphisms in type 1 diabetes: genetic association analysis and genotype-phenotype correlation in Chinese Han population. Autoimmunity. 2012;45(3):226-32. | CrossRef | Nejentsev S, Walker N, Riches D, Egholm M, Todd JA. Rare variants of IFIH1, a gene implicated in antiviral responses, protect against type 1 diabetes. Science. 2009;324(5925):387-9. | CrossRef |
Nejentsev S, Walker N, Riches D, Egholm M, Todd JA. Rare variants of IFIH1, a gene implicated in antiviral responses, protect against type 1 diabetes. Science. 2009;324(5925):387-9. | CrossRef | Rodríguez A, Alfaro JM, Balthazar V, Pineda Trujillo N. Association analysis of PTPN22, CTLA4 and IFIH1 genes with type 1 diabetes in Colombian families. J Diabetes. 2015;7(3):402-10. | CrossRef |
Rodríguez A, Alfaro JM, Balthazar V, Pineda Trujillo N. Association analysis of PTPN22, CTLA4 and IFIH1 genes with type 1 diabetes in Colombian families. J Diabetes. 2015;7(3):402-10. | CrossRef | Sadeghi R, Jahani F. Salting-in and salting-out of water-soluble polymers in aqueous salt solutions. J Phys Chem B. 2012;116(17):5234-41. | CrossRef |
Sadeghi R, Jahani F. Salting-in and salting-out of water-soluble polymers in aqueous salt solutions. J Phys Chem B. 2012;116(17):5234-41. | CrossRef | Kelsey J, Whittemore A, Evans A, Thompson W. Methods in Observational Epidemiology. Second edition ed. Oxford, UK: Oxford University Press;1996.
Kelsey J, Whittemore A, Evans A, Thompson W. Methods in Observational Epidemiology. Second edition ed. Oxford, UK: Oxford University Press;1996.  The International Genome Sample Resource. IGSR: The International Genome Sample Resource. 2018. | Link |
The International Genome Sample Resource. IGSR: The International Genome Sample Resource. 2018. | Link | Medina-Trujillo D. Estudio epidemiológico de los pacientes con diagnóstico de psoriasis en el Centro Dermatológico Federico Lleras Acosta (CDFLLA) del año 2011 a 2013. Bogotá, D.C.: Universidad Nacional de Colombia, Facultad de Medicina, Departamento de Medicina Interna, Programa de Dermatología; 2016. | Link |
Medina-Trujillo D. Estudio epidemiológico de los pacientes con diagnóstico de psoriasis en el Centro Dermatológico Federico Lleras Acosta (CDFLLA) del año 2011 a 2013. Bogotá, D.C.: Universidad Nacional de Colombia, Facultad de Medicina, Departamento de Medicina Interna, Programa de Dermatología; 2016. | Link | Parisi R, Symmons DP, Griffiths CE, Ashcroft DM, team IaMoPaACIp. Global epidemiology of psoriasis: a systematic review of incidence and prevalence. J Invest Dermatol. 2013;133(2):377-85. | CrossRef |
Parisi R, Symmons DP, Griffiths CE, Ashcroft DM, team IaMoPaACIp. Global epidemiology of psoriasis: a systematic review of incidence and prevalence. J Invest Dermatol. 2013;133(2):377-85. | CrossRef | Fernández-Armenteros JM, Gómez-Arbonés X, Buti-Solé M, Betriu-Bars A, Sanmartin-Novell V, Ortega-Bravo M, et al. Epidemiology of Psoriasis. A Population-Based Study. Actas Dermosifiliogr. 2018;110(5):385-392. | CrossRef |
Fernández-Armenteros JM, Gómez-Arbonés X, Buti-Solé M, Betriu-Bars A, Sanmartin-Novell V, Ortega-Bravo M, et al. Epidemiology of Psoriasis. A Population-Based Study. Actas Dermosifiliogr. 2018;110(5):385-392. | CrossRef | Chularojanamontri L, Kulthanan K, Suthipinittharm P, Jiamton S, Wongpraparut C, Silpa-Archa N, et al. Clinical differences between early- and late-onset psoriasis in Thai patients. Int J Dermatol. 2015;54(3):290-4. | CrossRef |
Chularojanamontri L, Kulthanan K, Suthipinittharm P, Jiamton S, Wongpraparut C, Silpa-Archa N, et al. Clinical differences between early- and late-onset psoriasis in Thai patients. Int J Dermatol. 2015;54(3):290-4. | CrossRef | Tan ES, Chong WS, Tey HL. Nail psoriasis: a review. Am J Clin Dermatol. 2012;13(6):375-88. | CrossRef |
Tan ES, Chong WS, Tey HL. Nail psoriasis: a review. Am J Clin Dermatol. 2012;13(6):375-88. | CrossRef | Alinaghi F, Calov M, Kristensen LE, Gladman DD, Coates LC, Jullien D, et al. Prevalence of psoriatic arthritis in patients with psoriasis: A systematic review and meta-analysis of observational and clinical studies. J Am Acad Dermatol. 2019;80(1):251-65.e19. | CrossRef |
Alinaghi F, Calov M, Kristensen LE, Gladman DD, Coates LC, Jullien D, et al. Prevalence of psoriatic arthritis in patients with psoriasis: A systematic review and meta-analysis of observational and clinical studies. J Am Acad Dermatol. 2019;80(1):251-65.e19. | CrossRef | Ray-Jones H, Eyre S, Barton A, Warren RB. One SNP at a Time: Moving beyond GWAS in Psoriasis. J Invest Dermatol. 2016;136(3):567-73. | CrossRef |
Ray-Jones H, Eyre S, Barton A, Warren RB. One SNP at a Time: Moving beyond GWAS in Psoriasis. J Invest Dermatol. 2016;136(3):567-73. | CrossRef | Zhang XJ, Huang W, Yang S, Sun LD, Zhang FY, Zhu QX, et al. Psoriasis genome-wide association study identifies susceptibility variants within LCE gene cluster at 1q21. Nat Genet. 2009;41(2):205-10. | CrossRef |
Zhang XJ, Huang W, Yang S, Sun LD, Zhang FY, Zhu QX, et al. Psoriasis genome-wide association study identifies susceptibility variants within LCE gene cluster at 1q21. Nat Genet. 2009;41(2):205-10. | CrossRef | Strange A, Capon F, Spencer CC, Knight J, Weale ME, Allen MH, et al. A genome-wide association study identifies new psoriasis susceptibility loci and an interaction between HLA-C and ERAP1. Nat Genet. 2010;42(11):985-90. | CrossRef |
Strange A, Capon F, Spencer CC, Knight J, Weale ME, Allen MH, et al. A genome-wide association study identifies new psoriasis susceptibility loci and an interaction between HLA-C and ERAP1. Nat Genet. 2010;42(11):985-90. | CrossRef | Yin X, Low HQ, Wang L, Li Y, Ellinghaus E, Han J, et al. Genome-wide meta-analysis identifies multiple novel associations and ethnic heterogeneity of psoriasis susceptibility. Nat Commun. 2015;6:6916. | CrossRef |
Yin X, Low HQ, Wang L, Li Y, Ellinghaus E, Han J, et al. Genome-wide meta-analysis identifies multiple novel associations and ethnic heterogeneity of psoriasis susceptibility. Nat Commun. 2015;6:6916. | CrossRef | van Vugt LJ, van den Reek JMPA, Coenen MJH, de Jong EMGJ. A systematic review of pharmacogenetic studies on the response to biologics in patients with psoriasis. Br J Dermatol. 2018;178(1):86-94. | CrossRef |
van Vugt LJ, van den Reek JMPA, Coenen MJH, de Jong EMGJ. A systematic review of pharmacogenetic studies on the response to biologics in patients with psoriasis. Br J Dermatol. 2018;178(1):86-94. | CrossRef |Systematization of initiatives in sexual and reproductive health about good practices criteria in response to the COVID-19 pandemic in primary health care in Chile
Clinical, psychological, social, and family characterization of suicidal behavior in Chilean adolescents: a multiple correspondence analysis









